Pola osadzania mogą być używane jako dane wejściowe/predyktory funkcji w regresji w taki sam sposób, jak w klasyfikacji.
Z tego samouczka dowiesz się, jak używać warstw pól osadzania 64D jako danych wejściowych do analizy regresji wielokrotnej, która prognozuje biomasę nadziemną (AGB).
Misja Global Ecosystem Dynamics Investigation (GEDI) NASA zbiera pomiary LIDAR wzdłuż transektów naziemnych w rozdzielczości przestrzennej 30 m w odstępach 60 m. Użyjemy zbioru danych GEDI L4A Raster Aboveground Biomass Density, który zawiera szacunkowe wartości gęstości biomasy nadziemnej (AGBD) i będzie używany jako zmienna przewidywana w modelu regresji.
Wybierz region
Zacznijmy od zdefiniowania obszaru zainteresowań. Na potrzeby tego samouczka wybierzemy region w Zachodnich Ghatach w Indiach i zdefiniujemy wielokąt jako zmienną geometryczną. Możesz też użyć narzędzi do rysowania w edytorze kodu, aby narysować wielokąt wokół interesującego Cię regionu. Zostanie on zapisany jako zmienna geometryczna w sekcji Imports (Importy). Korzystamy też z mapy bazowej Satelita, która ułatwia lokalizowanie obszarów porośniętych roślinnością.
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

Ilustracja: wybieranie obszaru zainteresowania na potrzeby prognozowania biomasy nadziemnej
Wybierz okres
Wybierz rok, dla którego chcesz przeprowadzić regresję. Pamiętaj, że osadzanie obrazów satelitarnych odbywa się w interwałach rocznych, więc okres definiujemy na cały rok.
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
Przygotowywanie zbioru danych osadzania satelitarnego
Obrazy osadzone z 64 pasami satelitarnymi będą używane jako predyktor regresji. Wczytujemy zbiór danych Satellite Embedding i filtrujemy obrazy z wybranego roku i regionu.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
Obrazy osadzone w satelicie są dzielone na kafelki i wyświetlane w projekcji dla stref UTM kafelka. W rezultacie otrzymujemy wiele kafelków z osadzonymi obrazami satelitarnymi obejmujących interesujący nas region. Aby uzyskać pojedynczy obraz, musimy je połączyć w mozaikę. W Earth Engine mozaika obrazów wejściowych ma domyślną projekcję, czyli WGS84 ze skalą 1 stopnia. Mozaikę będziemy później agregować i przekształcać w dalszej części samouczka, dlatego warto zachować oryginalną projekcję. Informacje o projekcji możemy wyodrębnić z jednego z kafelków i ustawić je na mozaice za pomocą funkcji setDefaultProjection().
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
Przygotowywanie mozaiki GEDI L4A
Szacunki biomasy GEDI będą używane do trenowania naszego modelu regresji, dlatego przed ich użyciem musimy odfiltrować nieprawidłowe lub niewiarygodne dane GEDI. Stosujemy kilka masek, aby usunąć potencjalnie błędne pomiary.
- Usuń wszystkie pomiary, które nie spełniają wymagań jakościowych (l4_quality_flag = 0 i degrade_flag > 0).
- Usuń wszystkie pomiary z wysokim błędem względnym ('agbd_se' / 'agbd' > 50%).
- Usuń wszystkie pomiary na zboczach o nachyleniu powyżej 30% na podstawie cyfrowego modelu wysokościowego (DEM) Copernicus GLO-30.
Na koniec wybieramy wszystkie pozostałe pomiary z interesującego nas okresu i regionu i tworzymy mozaikę.
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

Ilustracja: przygotowane obserwacje biomasy GEDI
Ponowne próbkowanie i agregowanie danych wejściowych
Przed pobraniem próbek pikseli do wytrenowania modelu regresji ponownie próbkujemy i przekształcamy dane wejściowe do tej samej siatki pikseli. Pomiary GEDI mają dokładność poziomą +/- 9 m. Jest to problematyczne w przypadku dopasowywania wartości GEDI AGB do pikseli Satellite Embedding. Aby temu zapobiec, ponownie próbkujemy i agregujemy wszystkie obrazy wejściowe do większej siatki pikseli z wartościami średnimi z oryginalnych pikseli. Pomaga to również usuwać szumy z danych i tworzyć lepszy model uczenia maszynowego.
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
Przekształcanie i agregowanie pikseli jest kosztowną operacją, dlatego warto wyeksportować wynikowy obraz warstwowy jako zasób i używać go w kolejnych krokach. Pomoże to uniknąć błędów przekroczenia limitu czasu obliczeń lub przekroczenia pamięci użytkownika podczas pracy z dużymi regionami.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Uruchom zadanie eksportowania i poczekaj na jego zakończenie. Następnie importujemy zasób i kontynuujemy tworzenie modelu.
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
Wyodrębnianie cech trenowania
Mamy już dane wejściowe gotowe do wyodrębnienia cech treningowych. W modelu regresji używamy pasm osadzania satelitarnego jako zmiennych zależnych (predyktorów) i wartości GEDI AGBD jako zmiennej niezależnej (przewidywanej). Możemy wyodrębnić wartości pokrywające się w każdym pikselu i przygotować zbiór danych treningowych. Obraz GEDI jest w większości zamaskowany i zawiera wartości tylko w niewielkim podzbiorze pikseli. Jeśli użyjemy sample(), zwróci ona głównie puste wartości. Aby temu zapobiec, tworzymy pasmo klas na podstawie maski GEDI i używamy stratifiedSample(), aby mieć pewność, że próbkowanie odbywa się na pikselach bez maski.
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
Trenowanie modelu regresji
Możemy już wytrenować model. Wiele klasyfikatorów w Earth Engine może być używanych do klasyfikacji i regresji. Ponieważ chcemy przewidzieć wartość liczbową (zamiast klasy), możemy ustawić klasyfikator w trybie REGRESSION i trenować go na podstawie danych treningowych. Po wytrenowaniu modelu możemy porównać jego prognozę z wartościami wejściowymi i obliczyć średni błąd kwadratowy (rmse) oraz współczynnik korelacji r^2, aby sprawdzić skuteczność modelu.
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
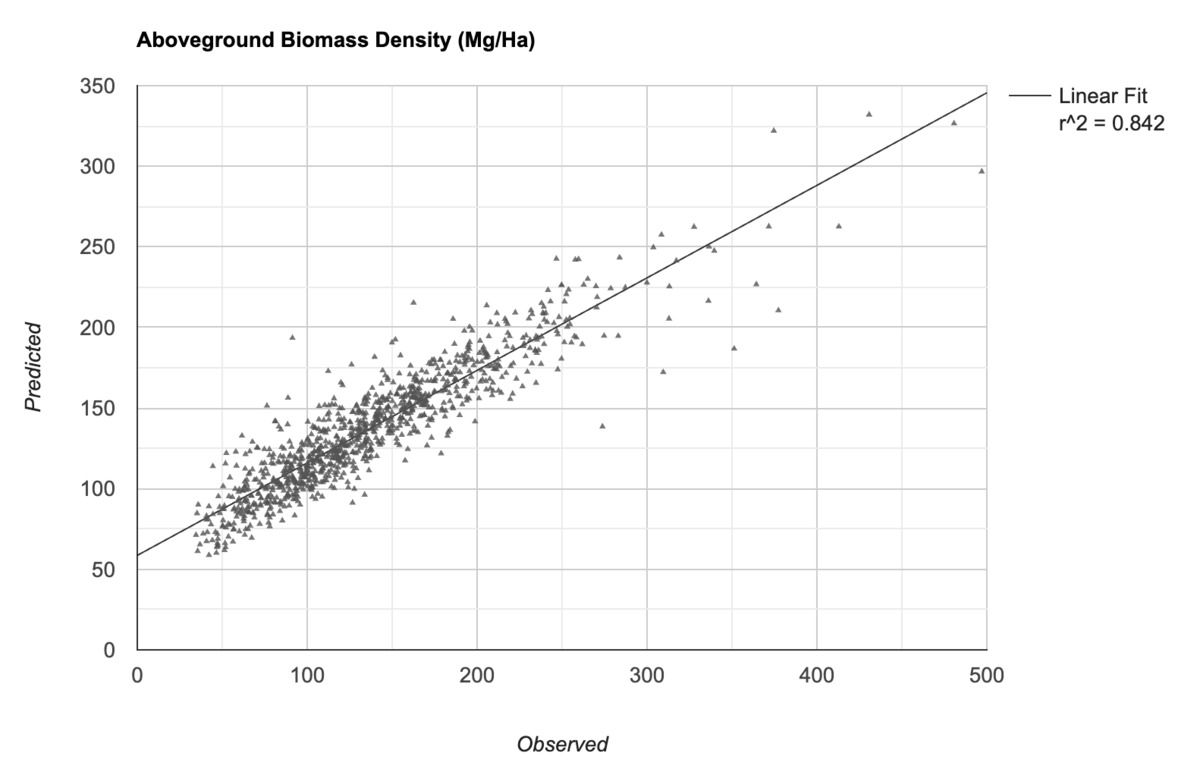
Ilustracja: rzeczywiste i prognozowane przez model wartości AGBD
Generowanie prognoz dla nieznanych wartości
Gdy będziemy zadowoleni z modelu, możemy go użyć do generowania prognoz w nieznanych lokalizacjach na podstawie obrazu zawierającego pasma predyktorów.
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
Obraz zawierający przewidywane wartości AGBD w każdym pikselu jest gotowy do wyeksportowania. Wykorzystamy to w następnej części, aby wizualizować wyniki.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Uruchom zadanie eksportowania i poczekaj na jego zakończenie. Po zakończeniu importujemy komponent i wyświetlamy wyniki.
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

Ilustracja: przewidywana wartość AGBD. Ciemniejszy zielony kolor oznacza większą przewidywaną gęstość biomasy.
Szacowanie łącznej biomasy
Mamy teraz przewidywane wartości AGBD dla każdego piksela obrazu, które można wykorzystać do oszacowania całkowitych zasobów biomasy nadziemnej (AGB) w regionie. Najpierw musimy jednak usunąć wszystkie piksele należące do obszarów bez roślinności. Możemy użyć zbioru danych dotyczących pokrycia terenu ESA WorldCover i wybrać piksele z roślinnością.
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

Ilustracja: prognozowana biomasa nadziemna z zamaskowanymi obszarami bez roślinności
Jednostką wartości GEDI AGBD są megagramy na hektar (Mg/ha). Aby uzyskać całkowitą wartość AGB, mnożymy każdy piksel przez jego powierzchnię w hektarach i sumujemy te wartości.
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);
Wypróbuj pełny skrypt z tego samouczka w edytorze kodu Earth Engine