Einbettungsfelder können als Feature-Eingaben/Vorhersagevariablen für die Regression verwendet werden, so wie sie für die Klassifizierung verwendet werden.
In dieser Anleitung erfahren Sie, wie Sie die 64D-Einbettungsfeld-Layer als Eingaben für eine multiple Regressionsanalyse verwenden, um die oberirdische Biomasse (Above-Ground Biomass, AGB) vorherzusagen.
Bei der Global Ecosystem Dynamics Investigation (GEDI) der NASA werden LIDAR-Messungen entlang von Bodentransekten mit einer räumlichen Auflösung von 30 m in Abständen von 60 m erfasst. Wir verwenden das Dataset GEDI L4A Raster Aboveground Biomass Density, das Punktschätzungen der oberirdischen Biomasse (Aboveground Biomass Density, AGBD) enthält, die als vorhergesagte Variable im Regressionsmodell verwendet werden.
Region auswählen
Beginnen wir mit der Definition einer Region of Interest. In diesem Beispiel wählen wir eine Region in den Western Ghats in Indien aus und definieren ein Polygon als Geometrie-Variable. Alternativ können Sie mit den Zeichenwerkzeugen im Code-Editor ein Polygon um den gewünschten Bereich zeichnen. Dieses wird dann als Geometrievariable in den Importen gespeichert. Wir verwenden auch die Satelliten-Basiskarte, mit der sich vegetationsreiche Gebiete leicht finden lassen.
var geometry = ee.Geometry.Polygon([[
[74.322, 14.981],
[74.322, 14.765],
[74.648, 14.765],
[74.648, 14.980]
]]);
// Use the satellite basemap
Map.setOptions('SATELLITE');

Abbildung: Auswahl des Interessenbereichs für die Vorhersage der oberirdischen Biomasse
Zeitraum auswählen
Wählen Sie ein Jahr aus, für das die Regression ausgeführt werden soll. Satelliteneinbettungen werden in Jahresintervallen zusammengefasst. Daher definieren wir den Zeitraum für das gesamte Jahr.
var startDate = ee.Date.fromYMD(2022, 1, 1);
var endDate = startDate.advance(1, 'year');
Dataset für Satelliteneinbettungen vorbereiten
Die 64‑Band-Satelliten-Embedding-Bilder werden als Vorhersagevariable für die Regression verwendet. Wir laden den Datensatz „Satellite Embedding“ und filtern nach Bildern für das ausgewählte Jahr und die ausgewählte Region.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var embeddingsFiltered = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
Bilder für die Einbettung von Satelliten werden in Kacheln unterteilt und in der Projektion für die UTM-Zonen der Kachel bereitgestellt. So erhalten wir mehrere Satellite Embedding-Kacheln, die die gewünschte Region abdecken. Um ein einzelnes Bild zu erhalten, müssen wir sie mosaikartig zusammenfügen. In Earth Engine wird einem Mosaik von Eingabe-Images die Standardprojektion zugewiesen, die WGS84 mit einem Maßstab von 1 Grad ist. Da wir dieses Mosaik später in der Anleitung aggregieren und neu projizieren, ist es hilfreich, die ursprüngliche Projektion beizubehalten. Wir können die Projektionsinformationen aus einer der Kacheln extrahieren und mit der Funktion setDefaultProjection() auf das Mosaik anwenden.
// Extract the projection of the first band of the first image
var embeddingsProjection = ee.Image(embeddingsFiltered.first()).select(0).projection();
// Set the projection of the mosaic to the extracted projection
var embeddingsImage = embeddingsFiltered.mosaic()
.setDefaultProjection(embeddingsProjection);
GEDI L4A-Mosaic vorbereiten
Da die GEDI-Biomasseschätzungen zum Trainieren unseres Regressionsmodells verwendet werden, ist es wichtig, ungültige oder unzuverlässige GEDI-Daten herauszufiltern, bevor sie verwendet werden. Wir wenden mehrere Masken an, um potenziell fehlerhafte Messungen zu entfernen.
- Entfernen Sie alle Messungen, die die Qualitätsanforderungen nicht erfüllen (l4_quality_flag = 0 und degrade_flag > 0).
- Entfernen Sie alle Messungen mit einem hohen relativen Fehler („agbd_se“ / „agbd“ > 50%).
- Entfernen Sie alle Messungen an Hängen mit einer Neigung von mehr als 30% basierend auf dem Copernicus GLO-30 Digital Elevation Mode (DEM).
Schließlich wählen wir alle verbleibenden Messungen für den relevanten Zeitraum und die relevante Region aus und erstellen ein Mosaik.
var gedi = ee.ImageCollection('LARSE/GEDI/GEDI04_A_002_MONTHLY');
// Function to select the highest quality GEDI data
var qualityMask = function(image) {
return image.updateMask(image.select('l4_quality_flag').eq(1))
.updateMask(image.select('degrade_flag').eq(0));
};
// Function to mask unreliable GEDI measurements
// with a relative standard error > 50%
// agbd_se / agbd > 0.5
var errorMask = function(image) {
var relative_se = image.select('agbd_se')
.divide(image.select('agbd'));
return image.updateMask(relative_se.lte(0.5));
};
// Function to mask GEDI measurements on slopes > 30%
var slopeMask = function(image) {
// Use Copernicus GLO-30 DEM for calculating slope
var glo30 = ee.ImageCollection('COPERNICUS/DEM/GLO30');
var glo30Filtered = glo30
.filter(ee.Filter.bounds(geometry))
.select('DEM');
// Extract the projection
var demProj = glo30Filtered.first().select(0).projection();
// The dataset consists of individual images
// Create a mosaic and set the projection
var elevation = glo30Filtered.mosaic().rename('dem')
.setDefaultProjection(demProj);
// Compute the slope
var slope = ee.Terrain.slope(elevation);
return image.updateMask(slope.lt(30));
};
var gediFiltered = gedi
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var gediProjection = ee.Image(gediFiltered.first())
.select('agbd').projection();
var gediProcessed = gediFiltered
.map(qualityMask)
.map(errorMask)
.map(slopeMask);
var gediMosaic = gediProcessed.mosaic()
.select('agbd').setDefaultProjection(gediProjection);
// Visualize the GEDI Mosaic
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(gediMosaic, gediVis, 'GEDI L4A (Filtered)', false);

Abbildung: Vorbereitete GEDI-Biomassebeobachtungen
Eingaben neu berechnen und aggregieren
Bevor wir Pixel für das Training eines Regressionsmodells auswählen, führen wir eine Resampling- und Reprojektion der Eingaben auf dasselbe Pixelraster durch. GEDI-Messungen haben eine horizontale Genauigkeit von +/- 9 m. Das ist problematisch, wenn die AGB-Werte von GEDI mit Satellite Embedding-Pixeln abgeglichen werden. Um dieses Problem zu beheben, werden alle Eingabebilder in ein größeres Pixelraster mit Mittelwerten aus den Originalpixeln neu berechnet und zusammengefasst. Außerdem wird so Rauschen aus den Daten entfernt und ein besseres Modell für maschinelles Lernen erstellt.
// Choose the grid size and projection
var gridScale = 100;
var gridProjection = ee.Projection('EPSG:3857')
.atScale(gridScale);
// Create a stacked image with predictor and predicted variables
var stacked = embeddingsImage.addBands(gediMosaic);
// Set the resampling mode
var stacked = stacked.resample('bilinear');
// Aggregate pixels with 'mean' statistics
var stackedResampled = stacked
.reduceResolution({
reducer: ee.Reducer.mean(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// As larger GEDI pixels contain masked original
// pixels, it has a transparency mask.
// We update the mask to remove the transparency
var stackedResampled = stackedResampled
.updateMask(stackedResampled.mask().gt(0));
Das Umprojizieren und Aggregieren von Pixeln ist ein aufwendiger Vorgang. Es empfiehlt sich, das resultierende gestapelte Bild als Asset zu exportieren und das vorab berechnete Bild in den nachfolgenden Schritten zu verwenden. So lassen sich die Fehler Zeitüberschreitung bei der Berechnung oder Arbeitsspeicher des Nutzers überschritten vermeiden, wenn Sie mit großen Regionen arbeiten.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var mosaicExportImage = 'gedi_mosaic';
var mosaicExportImagePath = exportFolder + mosaicExportImage;
Export.image.toAsset({
image: stackedResampled.clip(geometry),
description: 'GEDI_Mosaic_Export',
assetId: mosaicExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Starten Sie den Exportvorgang und warten Sie, bis er abgeschlossen ist. Anschließend importieren wir das Asset und fahren mit der Erstellung des Modells fort.
// Use the exported asset
var stackedResampled = ee.Image(mosaicExportImagePath);
Trainingsfunktionen extrahieren
Wir haben unsere Eingabedaten für das Extrahieren von Trainings-Features vorbereitet. Wir verwenden die Satellite Embedding-Bänder als abhängige Variablen (Vorhersagevariablen) und die GEDI-AGBD-Werte als unabhängige Variable (vorhergesagt) im Regressionsmodell. Wir können die übereinstimmenden Werte an jedem Pixel extrahieren und unser Trainingsdataset vorbereiten. Unser GEDI-Bild ist größtenteils maskiert und enthält nur für eine kleine Teilmenge von Pixeln Werte. Wenn wir sample() verwenden, werden meistens leere Werte zurückgegeben. Um dieses Problem zu beheben, erstellen wir ein Klassenband aus der GEDI-Maske und verwenden stratifiedSample(), um sicherzustellen, dass wir aus den nicht maskierten Pixeln Stichproben ziehen.
var predictors = embeddingsImage.bandNames();
var predicted = gediMosaic.bandNames().get(0);
print('predictors', predictors);
print('predicted', predicted);
var predictorImage = stackedResampled.select(predictors);
var predictedImage = stackedResampled.select([predicted]);
var classMask = predictedImage.mask().toInt().rename('class');
var numSamples = 1000;
// We set classPoints to [0, numSamples]
// This will give us 0 points for class 0 (masked areas)
// and numSample points for class 1 (non-masked areas)
var training = stackedResampled.addBands(classMask)
.stratifiedSample({
numPoints: numSamples,
classBand: 'class',
region: geometry,
scale: gridScale,
classValues: [0, 1],
classPoints: [0, numSamples],
dropNulls: true,
tileScale: 16,
});
print('Number of Features Extracted', training.size());
print('Sample Training Feature', training.first());
Regressionsmodell trainieren
Wir können das Modell jetzt trainieren. Viele Klassifizierer in Earth Engine können sowohl für Klassifizierungs- als auch für Regressionsaufgaben verwendet werden. Da wir einen numerischen Wert (und nicht eine Klasse) vorhersagen möchten, können wir den Klassifikator im Modus REGRESSION ausführen und mit den Trainingsdaten trainieren. Sobald das Modell trainiert ist, können wir die Vorhersage des Modells mit den Eingabewerten vergleichen und die Wurzel der mittleren Fehlerquadratsumme (rmse) und den Korrelationskoeffizienten r^2 berechnen, um die Leistung des Modells zu prüfen.
// Use the RandomForest classifier and set the
// output mode to REGRESSION
var model = ee.Classifier.smileRandomForest(50)
.setOutputMode('REGRESSION')
.train({
features: training,
classProperty: predicted,
inputProperties: predictors
});
// Get model's predictions for training samples
var predicted = training.classify({
classifier: model,
outputName: 'agbd_predicted'
});
// Calculate RMSE
var calculateRmse = function(input) {
var observed = ee.Array(
input.aggregate_array('agbd'));
var predicted = ee.Array(
input.aggregate_array('agbd_predicted'));
var rmse = observed.subtract(predicted).pow(2)
.reduce('mean', [0]).sqrt().get([0]);
return rmse;
};
var rmse = calculateRmse(predicted);
print('RMSE', rmse);
// Create a plot of observed vs. predicted values
var chart = ui.Chart.feature.byFeature({
features: predicted.select(['agbd', 'agbd_predicted']),
xProperty: 'agbd',
yProperties: ['agbd_predicted'],
}).setChartType('ScatterChart')
.setOptions({
title: 'Aboveground Biomass Density (Mg/Ha)',
dataOpacity: 0.8,
hAxis: {'title': 'Observed'},
vAxis: {'title': 'Predicted'},
legend: {position: 'right'},
series: {
0: {
visibleInLegend: false,
color: '#525252',
pointSize: 3,
pointShape: 'triangle',
},
},
trendlines: {
0: {
type: 'linear',
color: 'black',
lineWidth: 1,
pointSize: 0,
labelInLegend: 'Linear Fit',
visibleInLegend: true,
showR2: true
}
},
chartArea: {left: 100, bottom: 100, width: '50%'},
});
print(chart);
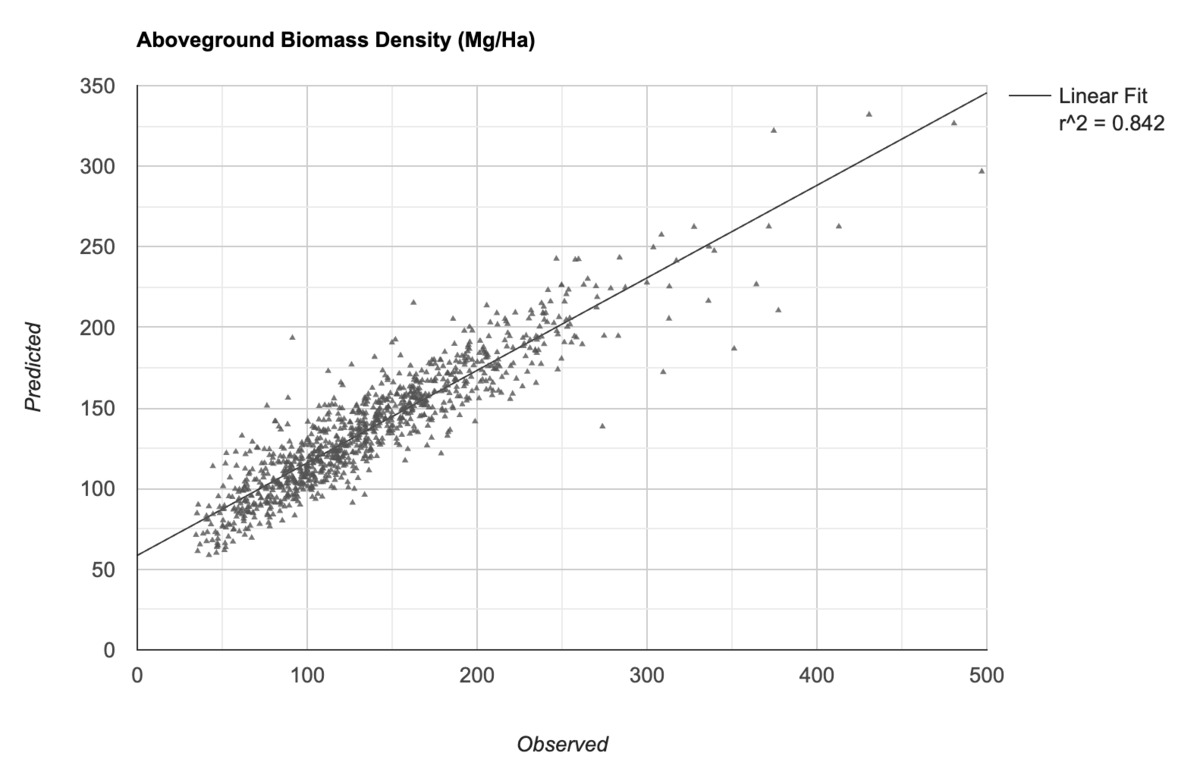
Abbildung: Beobachtete AGBD-Werte im Vergleich zu den vom Modell vorhergesagten Werten
Vorhersagen für unbekannte Werte generieren
Wenn wir mit dem Modell zufrieden sind, können wir es verwenden, um Vorhersagen für unbekannte Orte aus dem Bild mit den Vorhersagebändern zu generieren.
// We set the band name of the output image as 'agbd'
var predictedImage = stackedResampled.classify({
classifier: model,
outputName: 'agbd'
});
Das Bild mit den vorhergesagten AGBD-Werten für jedes Pixel kann jetzt exportiert werden. Wir verwenden diese Daten im nächsten Teil, um die Ergebnisse zu visualisieren.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var predictedExportImage = 'predicted_agbd';
var predictedExportImagePath = exportFolder + predictedExportImage;
Export.image.toAsset({
image: predictedImage.clip(geometry),
description: 'Predicted_Image_Export',
assetId: predictedExportImagePath,
region: geometry,
scale: gridScale,
maxPixels: 1e10
});
Starten Sie den Exportvorgang und warten Sie, bis er abgeschlossen ist. Anschließend importieren wir das Asset und visualisieren die Ergebnisse.
var predictedImage = ee.Image(predictedExportImagePath);
// Visualize the image
var gediVis = {
min: 0,
max: 200,
palette: ['#edf8fb', '#b2e2e2', '#66c2a4', '#2ca25f', '#006d2c'],
bands: ['agbd']
};
Map.addLayer(predictedImage, gediVis, 'Predicted AGBD');

Abbildung: Vorhersage für AGBD. Dunklere Grüntöne weisen auf eine höhere prognostizierte Biomasse-Dichte hin.
Gesamtbiomasse schätzen
Wir haben jetzt geschätzte AGBD-Werte für jeden Pixel des Bildes, die verwendet werden können, um den gesamten oberirdischen Biomassebestand (AGB) in der Region zu schätzen. Zuerst müssen wir jedoch alle Pixel entfernen, die zu nicht bewachsenen Gebieten gehören. Wir können den ESA WorldCover-Datensatz zur Landbedeckung verwenden und vegetationsbedeckte Pixel auswählen.
// GEDI data is processed only for certain landcovers
// from Plant Functional Types (PFT) classification
// https://doi.org/10.1029/2022EA002516
// Here we use ESA WorldCover v200 product to
// select landcovers representing vegetated areas
var worldcover = ee.ImageCollection('ESA/WorldCover/v200').first();
// Aggregate pixels to the same grid as other dataset
// with 'mode' value.
// i.e. The landcover with highest occurrence within the grid
var worldcoverResampled = worldcover
.reduceResolution({
reducer: ee.Reducer.mode(),
maxPixels: 1024
})
.reproject({
crs: gridProjection
});
// Select grids for the following classes
// | Class Name | Value |
// | Forests | 10 |
// | Shrubland | 20 |
// | Grassland | 30 |
// | Cropland | 40 |
// | Mangroves | 95 |
var landCoverMask = worldcoverResampled.eq(10)
.or(worldcoverResampled.eq(20))
.or(worldcoverResampled.eq(30))
.or(worldcoverResampled.eq(40))
.or(worldcoverResampled.eq(95));
var predictedImageMasked = predictedImage
.updateMask(landCoverMask);
Map.addLayer(predictedImageMasked, gediVis, 'Predicted AGBD (Masked)');

Abbildung: Vorhersage der AGBD mit maskierten nicht vegetationsbedeckten Bereichen
Die Einheiten der GEDI-AGBD-Werte sind Megagramm pro Hektar (Mg/ha). Um die gesamte AGB zu erhalten, multiplizieren wir jeden Pixel mit seiner Fläche in Hektar und addieren die Werte.
var pixelAreaHa = ee.Image.pixelArea().divide(10000);
var predictedAgb = predictedImageMasked.multiply(pixelAreaHa);
var stats = predictedAgb.reduceRegion({
reducer: ee.Reducer.sum(),
geometry: geometry,
scale: gridScale,
maxPixels: 1e10,
tileScale: 16
});
// Result is a dictionary with key for each band
var totalAgb = stats.getNumber('agbd');
print('Total AGB (Mg)', totalAgb);
Vollständiges Skript für diese Anleitung im Earth Engine-Code-Editor ausprobieren