Im vorherigen Tutorial (Einführung) haben wir gesehen, wie mit Satellite Embeddings jährliche Trajektorien von Satellitenbeobachtungen und Klimavariablen erfasst werden. Dadurch ist das Dataset sofort für die Kartierung von Feldfrüchten nutzbar, ohne dass die Phänologie der Feldfrüchte modelliert werden muss. Die Zuordnung von Erntetypen ist eine anspruchsvolle Aufgabe, die in der Regel die Modellierung der Pflanzenphänologie und die Sammlung von Feldproben für alle in der Region angebauten Pflanzen erfordert.
In dieser Anleitung verwenden wir einen Ansatz für die unüberwachte Klassifizierung für die Kartierung von Feldfrüchten, mit dem wir diese komplexe Aufgabe ohne Feldlabels ausführen können. Bei dieser Methode werden lokales Wissen über die Region sowie aggregierte Erntestatistiken genutzt, die für viele Teile der Welt leicht verfügbar sind.
Region auswählen
In dieser Anleitung erstellen wir eine Karte mit Erntetypen für Cerro Gordo County, Iowa. Dieser Bezirk liegt im Corn Belt der USA, wo hauptsächlich Mais und Sojabohnen angebaut werden. Dieses lokale Wissen ist wichtig und hilft uns, wichtige Parameter für unser Modell festzulegen.
Zuerst müssen wir die Grenze für den ausgewählten Bezirk abrufen.
// Select the region
// Cerro Gordo County, Iowa
var counties = ee.FeatureCollection('TIGER/2018/Counties');
var selected = counties
.filter(ee.Filter.eq('GEOID', '19033'));
var geometry = selected.geometry();
Map.centerObject(geometry, 12);
Map.addLayer(geometry, {color: 'red'}, 'Selected Region', false);

Abbildung: Ausgewählte Region
Dataset für Satelliteneinbettungen vorbereiten
Als Nächstes laden wir das Satellite Embedding-Dataset, filtern nach Bildern für das ausgewählte Jahr und erstellen ein Mosaik.
var embeddings = ee.ImageCollection('GOOGLE/SATELLITE_EMBEDDING/V1/ANNUAL');
var year = 2022;
var startDate = ee.Date.fromYMD(year, 1, 1);
var endDate = startDate.advance(1, 'year');
var filteredembeddings = embeddings
.filter(ee.Filter.date(startDate, endDate))
.filter(ee.Filter.bounds(geometry));
var embeddingsImage = filteredembeddings.mosaic();
Anbaumasken erstellen
Für unsere Modellierung müssen wir Nicht-Ackerland ausschließen. Es gibt viele globale und regionale Datasets, die zum Erstellen einer Zuschneidemaske verwendet werden können. ESA WorldCover oder GFSAD Global Cropland Extent Product sind gute Optionen für globale Datasets zu Ackerland. Eine neuere Ergänzung ist das Produkt ESA WorldCereal Active Cropland, das saisonale Karten von aktiven Ackerflächen enthält. Da sich unsere Region in den USA befindet, können wir einen genaueren regionalen Datensatz verwenden, nämlich die USDA NASS Cropland Data Layers (CDL), um eine Erntemaske zu erhalten.
// Use Cropland Data Layers (CDL) to obtain cultivated cropland
var cdl = ee.ImageCollection('USDA/NASS/CDL')
.filter(ee.Filter.date(startDate, endDate))
.first();
var cropLandcover = cdl.select('cropland');
var croplandMask = cdl.select('cultivated').eq(2).rename('cropmask');
// Visualize the crop mask
var croplandMaskVis = {min: 0, max: 1, palette: ['white', 'green']};
Map.addLayer(croplandMask.clip(geometry), croplandMaskVis, 'Crop Mask');
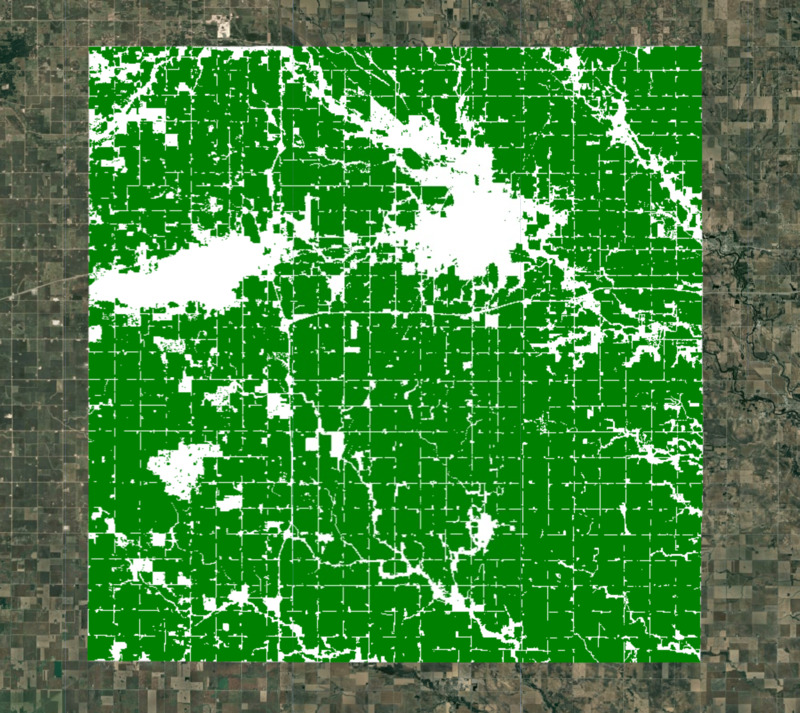
Abbildung: Ausgewählte Region mit Maske für landwirtschaftliche Flächen
Trainingsbeispiele extrahieren
Wir wenden die Maske für landwirtschaftliche Flächen auf das Einbettungs-Mosaik an. Übrig bleiben alle Pixel, die landwirtschaftliche Nutzflächen im Landkreis darstellen.
// Mask all non-cropland pixels
var clusterImage = embeddingsImage.updateMask(croplandMask);
Wir benötigen das Satelliten-Embedding-Bild und müssen Zufallsstichproben nehmen, um ein Clustering-Modell zu trainieren. Da unsere Region of Interest viele maskierte Pixel enthält, kann es bei einer einfachen Zufallsstichprobe zu Stichproben mit Nullwerten kommen. Damit wir die gewünschte Anzahl von Stichproben ohne Nullwerte extrahieren können, verwenden wir die geschichtete Stichprobenerhebung, um die gewünschte Anzahl von Stichproben in nicht maskierten Bereichen zu erhalten.
// Stratified random sampling
var training = clusterImage.addBands(croplandMask).stratifiedSample({
numPoints: 1000,
classBand: 'cropmask',
region: geometry,
scale: 10,
tileScale: 16,
seed: 100,
dropNulls: true,
geometries: true
});
Beispiel in ein Asset exportieren (optional)
Das Extrahieren von Beispielen ist ein rechenintensiver Vorgang. Es empfiehlt sich, die extrahierten Trainingsbeispiele als Assets zu exportieren und die exportierten Assets in den nachfolgenden Schritten zu verwenden. So lassen sich die Fehler Zeitüberschreitung bei der Berechnung oder Arbeitsspeicher des Nutzers überschritten bei der Arbeit mit großen Regionen vermeiden.
Starten Sie den Exportvorgang und warten Sie, bis er abgeschlossen ist, bevor Sie fortfahren.
// Replace this with your asset folder
// The folder must exist before exporting
var exportFolder = 'projects/spatialthoughts/assets/satellite_embedding/';
var samplesExportFc = 'cluster_training_samples';
var samplesExportFcPath = exportFolder + samplesExportFc;
Export.table.toAsset({
collection: training,
description: 'Cluster_Training_Samples',
assetId: samplesExportFcPath
});
Nachdem der Exportvorgang abgeschlossen ist, können wir die extrahierten Stichproben als Feature-Sammlung wieder in unseren Code einlesen.
// Use the exported asset
var training = ee.FeatureCollection(samplesExportFcPath);
Beispiele visualisieren
Unabhängig davon, ob Sie die Stichprobe interaktiv ausgeführt oder in eine Feature-Sammlung exportiert haben, verfügen Sie jetzt über eine Trainingsvariable mit Ihren Stichprobenpunkten. Lassen Sie uns das erste Beispiel ausgeben, um es zu prüfen und unsere Trainingspunkte der Map hinzuzufügen.
print('Extracted sample', training.first());
Map.addLayer(training, {color: 'blue'}, 'Extracted Samples', false);

Abbildung: Extrahierte Zufallsstichproben für das Clustering
Unüberwachtes Clustering durchführen
Wir können jetzt einen Clusterer trainieren und die 64D-Einbettungsvektoren in eine ausgewählte Anzahl von unterschiedlichen Clustern gruppieren. Unseren Ortskenntnissen zufolge gibt es zwei Hauptkulturen, die den Großteil der Fläche ausmachen. Auf dem verbleibenden Teil werden mehrere andere Kulturen angebaut. Wir können unüberwachtes Clustering für das Satellite Embedding durchführen, um Cluster von Pixeln mit ähnlichen zeitlichen Verläufen und Mustern zu erhalten. Pixel mit ähnlichen spektralen und räumlichen Eigenschaften sowie ähnlicher Phänologie werden in derselben Gruppe zusammengefasst.
Mit ee.Clusterer.wekaCascadeKMeans() können wir eine Mindest- und eine Höchstanzahl von Clustern angeben und die optimale Anzahl von Clustern anhand der Trainingsdaten ermitteln. Hier ist unser lokales Wissen hilfreich, um die Mindest- und Höchstzahl von Clustern festzulegen. Da wir einige unterschiedliche Arten von Clustern erwarten – Mais, Sojabohnen und mehrere andere Nutzpflanzen – können wir 4 als Mindestanzahl von Clustern und 5 als Höchstanzahl von Clustern verwenden. Möglicherweise müssen Sie mit diesen Zahlen experimentieren, um herauszufinden, was für Ihre Region am besten funktioniert.
// Cluster the Satellite Embedding Image
var minClusters = 4;
var maxClusters = 5;
var clusterer = ee.Clusterer.wekaCascadeKMeans({
minClusters: minClusters, maxClusters: maxClusters}).train({
features: training,
inputProperties: clusterImage.bandNames()
});
var clustered = clusterImage.cluster(clusterer);
Map.addLayer(clustered.randomVisualizer().clip(geometry), {}, 'Clusters');
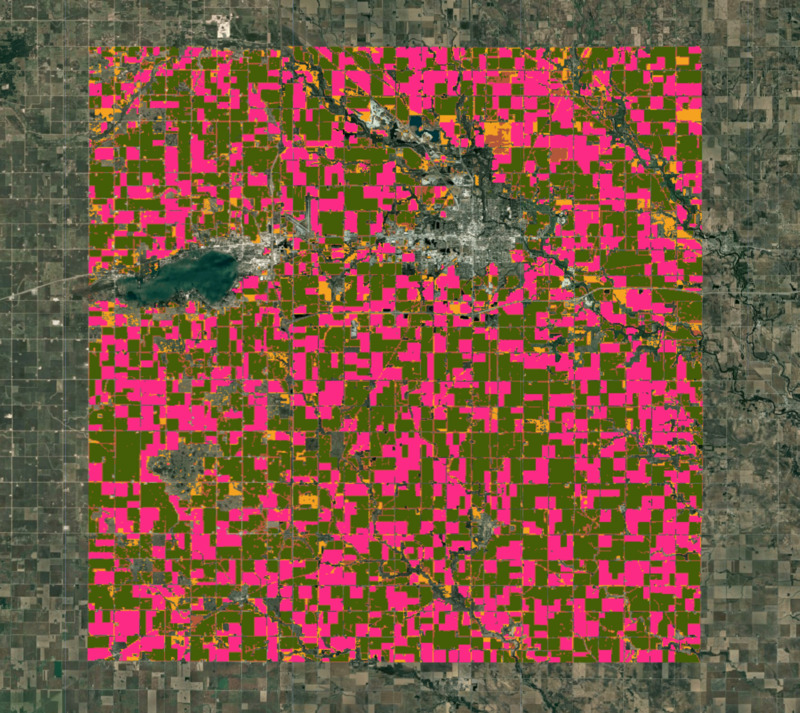
Abbildung: Cluster aus unüberwachter Klassifizierung
Clustern Labels zuweisen
Bei der visuellen Prüfung stimmen die in den vorherigen Schritten erhaltenen Cluster genau mit den Grenzen der Farm auf dem hochauflösenden Bild überein. Wir wissen aus lokaler Erfahrung, dass die beiden größten Cluster Mais und Sojabohnen sind. Berechnen wir die Flächen der einzelnen Cluster in unserem Bild.
// Calculate Cluster Areas
// 1 Acre = 4046.86 Sq. Meters
var areaImage = ee.Image.pixelArea().divide(4046.86).addBands(clustered);
var areas = areaImage.reduceRegion({
reducer: ee.Reducer.sum().group({
groupField: 1,
groupName: 'cluster',
}),
geometry: geometry,
scale: 10,
maxPixels: 1e10
});
var clusterAreas = ee.List(areas.get('groups'));
// Process results to extract the areas and create a FeatureCollection
var clusterAreas = clusterAreas.map(function(item) {
var areaDict = ee.Dictionary(item);
var clusterNumber = areaDict.getNumber('cluster').format();
var area = areaDict.getNumber('sum');
return ee.Feature(null, {cluster: clusterNumber, area: area});
});
var clusterAreaFc = ee.FeatureCollection(clusterAreas);
print('Cluster Areas', clusterAreaFc);
Wir wählen die beiden Cluster mit der größten Fläche aus.
var selectedFc = clusterAreaFc.sort('area', false).limit(2);
print('Top 2 Clusters by Area', selectedFc);
Wir wissen aber immer noch nicht, welche Cluster welche Kulturpflanze darstellen. Wenn Sie einige Feldproben von Mais oder Sojabohnen hätten, könnten Sie sie auf die Cluster legen, um die entsprechenden Labels zu ermitteln. Wenn keine Feldproben vorhanden sind, können wir aggregierte Erntestatistiken verwenden. In vielen Teilen der Welt werden regelmäßig aggregierte Erntestatistiken erhoben und veröffentlicht. In den USA bietet der National Agricultural Statistics Service (NASS) detaillierte Erntestatistiken für jeden Bezirk und jede wichtige Nutzpflanze. Im Jahr 2022 betrug die Anbaufläche für Mais in Cerro Gordo County, Iowa, 161.500 Acres und die Anbaufläche für Sojabohnen 110.500 Acres.
Anhand dieser Informationen wissen wir nun, dass es sich bei dem Cluster mit der größten Fläche mit hoher Wahrscheinlichkeit um Mais und beim anderen um Sojabohnen handelt. Weisen wir diese Labels zu und vergleichen wir die berechneten Flächen mit den veröffentlichten Statistiken.
var cornFeature = selectedFc.sort('area', false).first();
var soybeanFeature = selectedFc.sort('area').first();
var cornCluster = cornFeature.get('cluster');
var soybeanCluster = soybeanFeature.get('cluster');
print('Corn Area (Detected)', cornFeature.getNumber('area').round());
print('Corn Area (From Crop Statistics)', 163500);
print('Soybean Area (Detected)', soybeanFeature.getNumber('area').round());
print('Soybean Area (From Crop Statistics)', 110500);
Erntekarte erstellen
Wir kennen jetzt die Labels für jeden Cluster und können die Pixel für jeden Erntetyp extrahieren und zusammenführen, um die endgültige Erntekarte zu erstellen.
// Select the clusters to create the crop map
var corn = clustered.eq(ee.Number.parse(cornCluster));
var soybean = clustered.eq(ee.Number.parse(soybeanCluster));
var merged = corn.add(soybean.multiply(2));
var cropVis = {min: 0, max: 2, palette: ['#bdbdbd', '#ffd400', '#267300']};
Map.addLayer(merged.clip(geometry), cropVis, 'Crop Map (Detected)');
Um die Ergebnisse besser interpretieren zu können, können wir auch UI-Elemente verwenden, um eine Legende für die Karte zu erstellen und hinzuzufügen.
// Add a Legend
var legend = ui.Panel({
layout: ui.Panel.Layout.Flow('horizontal'),
style: {position: 'bottom-center', padding: '8px 15px'}});
var addItem = function(color, name) {
var colorBox = ui.Label({
style: {color: '#ffffff',
backgroundColor: color,
padding: '10px',
margin: '0 4px 4px 0',
}
});
var description = ui.Label({
value: name,
style: {
margin: '0px 10px 0px 2px',
}
});
return ui.Panel({
widgets: [colorBox, description],
layout: ui.Panel.Layout.Flow('horizontal')
});
};
var title = ui.Label({
value: 'Legend',
style: {
fontWeight: 'bold',
fontSize: '16px',
margin: '0px 10px 0px 4px'
}
});
legend.add(title);
legend.add(addItem('#ffd400', 'Corn'));
legend.add(addItem('#267300', 'Soybean'));
legend.add(addItem('#bdbdbd', 'Other Crops'));
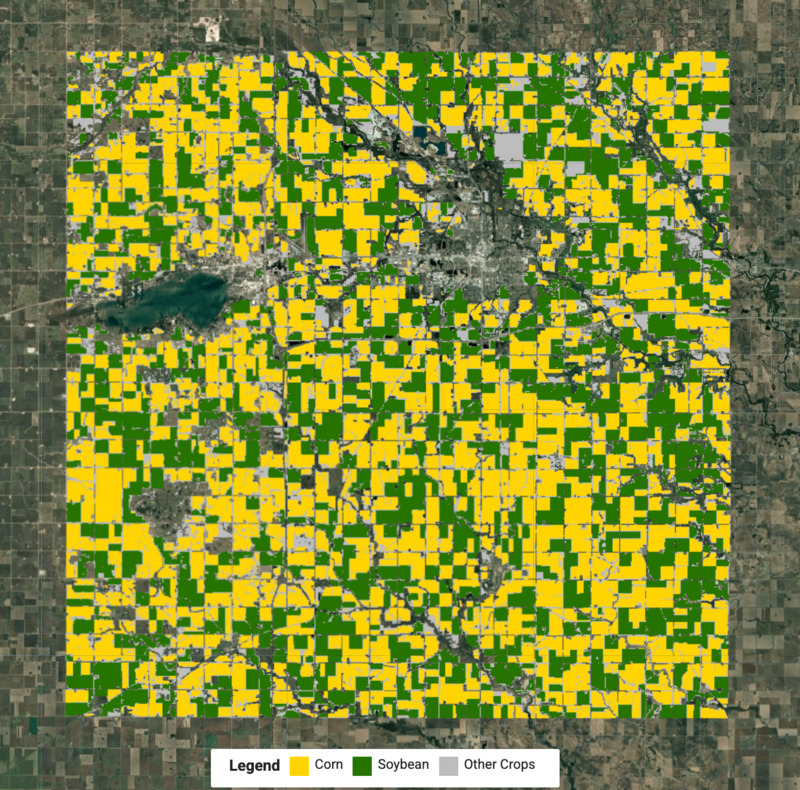
Abbildung: Erkannte Karte mit Mais- und Sojabohnenfeldern
Ergebnisse prüfen
Wir konnten eine Karte mit Erntetypen mit dem Satellite Embedding-Dataset ohne Feldlabels erstellen, indem wir nur die aggregierten Statistiken und Ortskenntnisse der Region verwendet haben. Vergleichen wir unsere Ergebnisse mit der offiziellen Karte der Kulturpflanzentypen aus den CDL-Daten (Cropland Data Layers) des USDA NASS (National Agricultural Statistics Service).
var cdl = ee.ImageCollection('USDA/NASS/CDL')
.filter(ee.Filter.date(startDate, endDate))
.first();
var cropLandcover = cdl.select('cropland');
var cropMap = cropLandcover.updateMask(croplandMask).rename('crops');
// Original data has unique values for each crop ranging from 0 to 254
var cropClasses = ee.List.sequence(0, 254);
// We remap all values as following
// Crop | Source Value | Target Value
// Corn | 1 | 1
// Soybean | 5 | 2
// All other| 0-255 | 0
var targetClasses = ee.List.repeat(0, 255).set(1, 1).set(5, 2);
var cropMapReclass = cropMap.remap(cropClasses, targetClasses).rename('crops');
var cropVis = {min: 0, max: 2, palette: ['#bdbdbd', '#ffd400', '#267300']};
Map.addLayer(cropMapReclass.clip(geometry), cropVis, 'Crop Landcover (CDL)');

Abbildung: (links) Karte aus Satelliteneinbettungen zuschneiden, (rechts) Karte aus CDL zuschneiden
Obwohl es Abweichungen zwischen unseren Ergebnissen und der offiziellen Karte gibt, haben wir mit minimalem Aufwand ziemlich gute Ergebnisse erzielt. Durch die Anwendung von Nachbearbeitungsschritten auf die Ergebnisse können wir Rauschen entfernen und Lücken in der Ausgabe schließen.
Vollständiges Skript für diese Anleitung im Earth Engine-Code-Editor ausprobieren