Bu eğiticide, Google Earth Engine kullanılarak tür dağılımı modelleme metodolojisi tanıtılacaktır. Öncelikle Tür Dağılımı Modellemesi hakkında kısa bir genel bakış sunulacak, ardından peri pitta (bilimsel adı: Pitta nympha) olarak bilinen nesli tükenmekte olan bir kuş türünün yaşam alanını tahmin etme ve analiz etme süreci anlatılacak.
Önce beni çalıştır
API'yi başlatmak için aşağıdaki hücreyi çalıştırın. Çıkışta, hesabınızı kullanarak bu not defterine Earth Engine erişimi verme talimatları yer alır.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
Tür Dağılımı Modellemesi'ne kısa bir genel bakış
Tür dağılımı modellerinin ne olduğunu, bu modellerin işlenmesi için Google Earth Engine'i kullanmanın avantajlarını, modeller için gereken verileri ve iş akışının nasıl yapılandırıldığını inceleyelim.
Tür Dağılımı Modellemesi nedir?
Tür Dağılımı Modellemesi (SDM), bir türün gerçek veya potansiyel coğrafi dağılımını tahmin etmek için kullanılan en yaygın yöntemdir. Belirli bir tür için uygun olan çevre koşullarını tanımlamayı ve ardından bu uygun koşulların coğrafi olarak nerede bulunduğunu belirlemeyi içerir.
SDM, son yıllarda koruma planlamasının önemli bir bileşeni olarak ortaya çıkmıştır ve bu amaçla çeşitli modelleme teknikleri geliştirilmiştir. Google Earth Engine'de (GEE) SDM'nin uygulanması, büyük ölçekli çevre verilerine kolay erişim, güçlü bilgi işlem özellikleri ve makine öğrenimi algoritmaları için destek sağlayarak hızlı modelleme olanağı sunar.
SDM için Gerekli Veriler
SDM, bir popülasyonun sürdürülebileceği koşulları belirlemek için genellikle bilinen türlerin oluşum kayıtları ile çevresel değişkenler arasındaki ilişkiden yararlanır. Diğer bir deyişle, iki tür model giriş verisi gerekir:
- Bilinen türlerin oluşum kayıtları
- Çeşitli çevre değişkenleri
Bu veriler, türlerin varlığıyla ilişkili çevresel koşulları belirlemek için algoritmalara girilir.
GEE ile SDM iş akışı
GEE kullanan SDM'nin iş akışı aşağıdaki gibidir:
- Tür oluşumu verilerinin toplanması ve ön işlenmesi
- Alakalı Konumun Tanımı
- GEE ortam değişkenlerinin eklenmesi
- Sözde yokluk verilerinin oluşturulması
- Model uyumu ve tahmin
- Değişken önemi ve doğruluk değerlendirmesi
GEE Kullanarak Habitat Tahmini ve Analizi
GEE tabanlı SDM'nin uygulanmasını göstermek için peri pitası (Pitta nympha) örnek olay olarak kullanılacaktır. Bu örnek için belirli bir tür seçilmiş olsa da araştırmacılar, sağlanan kaynak kodda küçük değişiklikler yaparak bu metodolojiyi ilgilendikleri herhangi bir hedef türe uygulayabilir.
Peri pitta, Güney Kore'de nadir görülen bir yaz göçmeni ve geçiş göçmenidir. Dağıtım alanı, Kore Yarımadası'ndaki son iklim ısınması nedeniyle genişlemektedir. Nadir bir tür, II. sınıf tehlike altındaki yaban hayatı, 204 numaralı doğal anıt olarak sınıflandırılır. Ulusal Kırmızı Liste'de Bölgesel Olarak Nesli Tükenmiş (RE), IUCN kategorilerine göre ise Hassas (VU) olarak değerlendirilir.
Peri pitasının korunması için planlama yapılırken SDM'nin uygulanması oldukça faydalı olacaktır. Şimdi de GEE aracılığıyla yaşam alanı tahmini ve analizine geçelim.
Öncelikle Python kitaplıkları içe aktarılır.import ifadesi bir modülün tüm içeriğini getirirken from import ifadesi, bir modülden belirli nesnelerin içe aktarılmasına olanak tanır.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
Tür oluşumu verilerinin toplanması ve önceden işlenmesi
Şimdi de peri pitta kuşu için oluşum verilerini toplayalım. İlgilendiğiniz türlerin oluşum verilerine şu anda erişiminiz olmasa bile GBIF API aracılığıyla belirli türler hakkında gözlem verileri edinebilirsiniz. GBIF API, GBIF tarafından sağlanan tür dağılımı verilerine erişim sağlayan bir arayüzdür. Kullanıcıların verileri aramasına, filtrelemesine ve indirmesine, ayrıca türlerle ilgili çeşitli bilgiler edinmesine olanak tanır.
Aşağıdaki kodda, species_name değişkenine türün bilimsel adı atanır (ör. Pitta nympha (Peri pitası için) ve country_code değişkenine ülke kodu atanır (ör. Güney Kore için KR). base_url değişkeni, GBIF API'sinin adresini depolar. params, API isteğinde kullanılacak parametreleri içeren bir sözlüktür:
scientificName: Aranacak türün bilimsel adını ayarlar.country: Aramayı belirli bir ülkeyle sınırlar.hasCoordinate: Yalnızca koordinat içeren verilerin (doğru) aranmasını sağlar.basisOfRecord: Yalnızca insan gözlemine ait kayıtları seçer (HUMAN_OBSERVATION).limit: Döndürülen sonuçların maksimum sayısını 10.000 olarak ayarlar.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
Daha önce ayarlanan parametreleri kullanarak GBIF API'sinde peri pitası (Pitta nympha) ile ilgili gözlem kayıtlarını sorguluyoruz ve sonuçları ilk satırı kontrol etmek için bir DataFrame'e yüklüyoruz. DataFrame, satır ve sütunlardan oluşan, tablo biçimindeki verileri işlemek için kullanılan bir veri yapısıdır. Gerekirse DataFrame, CSV dosyası olarak kaydedilip tekrar okunabilir.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
Ardından, DataFrame'i coğrafi bilgiler için bir sütun içeren bir GeoDataFrame'e dönüştürüp (geometry) ilk satırı kontrol ediyoruz. GeoDataFrame, GeoPackage dosyası (*.gpkg) olarak kaydedilebilir ve tekrar okunabilir.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
Bu kez, GeoDataFrame'deki verilerin yıla ve aya göre dağılımını görselleştirmek ve bunu grafik olarak göstermek için bir işlev oluşturduk. Bu grafik daha sonra resim dosyası olarak kaydedilebilir. Isı haritası kullanımı, türlerin görülme sıklığını yıla ve aya göre hızlı bir şekilde anlamamıza olanak tanır. Bu sayede, verilerdeki zamansal değişikliklerin ve kalıpların sezgisel bir görselleştirmesini sunar. Bu sayede, tür oluşumu verilerindeki zamansal kalıplar ve mevsimsel değişiklikler belirlenebilir, ayrıca verilerdeki aykırı değerler veya kalite sorunları hızlıca tespit edilebilir.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
1995'teki veriler çok seyrek olup diğer yıllara kıyasla önemli boşluklar vardır. Ayrıca Ağustos ve Eylül aylarında da sınırlı örnekler bulunur ve diğer dönemlere kıyasla farklı mevsimsel özellikler görülür. Bu verilerin hariç tutulması, modelin kararlılığının ve tahmin gücünün artırılmasına katkıda bulunabilir.
Ancak verilerin hariç tutulmasının modelin genelleme yeteneğini artırabileceğini, ancak araştırma hedefleriyle ilgili değerli bilgilerin kaybolmasına da yol açabileceğini unutmamak önemlidir. Bu nedenle, bu tür kararlar dikkatli bir şekilde verilmelidir.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
Filtrelenmiş GeoDataFrame, Google Earth Engine nesnesine dönüştürülür.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
Ardından, SDM sonuçlarının raster piksel boyutunu 1 km çözünürlük olarak tanımlayacağız.
# Spatial resolution setting (meters)
grain_size = 1000
Aynı 1 km çözünürlüklü raster pikselde birden fazla oluşum noktası bulunduğunda, bu noktaların aynı coğrafi konumda aynı çevre koşullarını paylaşma olasılığı yüksektir. Bu tür verilerin doğrudan analizde kullanılması sonuçlara önyargı katabilir.
Başka bir deyişle, coğrafi örnekleme yanlılığının olası etkisini sınırlamamız gerekir. Bunu sağlamak için her 1 km'lik pikselde yalnızca bir konum tutacak ve diğer tüm konumları kaldıracağız. Böylece model, çevre koşullarını daha nesnel bir şekilde yansıtacak.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
Aşağıda, ön işleme öncesinde (mavi) ve sonrasında (kırmızı) coğrafi örnekleme yanlılığını karşılaştıran görselleştirme gösterilmektedir. Karşılaştırmayı kolaylaştırmak için harita, Hallasan Milli Parkı'nda yüksek oranda peri pitta koordinatlarının bulunduğu bölgeye göre ortalanmıştır.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
Alakalı Konumun Tanımı
İlgilenilen alanın (aşağıda AOI) tanımlanması, araştırmacıların analiz etmek istedikleri coğrafi alanı belirtmek için kullandıkları terimi ifade eder. Çalışma alanı terimiyle benzer bir anlama sahiptir.
Bu bağlamda, ilgi alanını tanımlamak için oluşum noktası katmanı geometrisinin sınırlayıcı kutusunu elde ettik ve etrafında 50 kilometrelik bir arabellek (maksimum 1.000 metre toleransla) oluşturduk.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
GEE ortam değişkenlerinin eklenmesi
Şimdi analize ortam değişkenleri ekleyelim. GEE; sıcaklık, yağış, yükseklik, arazi örtüsü ve arazi gibi çevresel değişkenler için çok çeşitli veri kümeleri sağlar. Bu veri kümeleri, peri pitasının habitat tercihlerini etkileyebilecek çeşitli faktörleri kapsamlı bir şekilde analiz etmemizi sağlar.
SDM'de GEE çevre değişkenlerinin seçimi, türün yaşam alanı tercihi özelliklerini yansıtmalıdır. Bunun için peri pitasının yaşam alanı tercihleri hakkında önceden araştırma ve literatür taraması yapılması gerekir. Bu eğitimde öncelikle GEE kullanılarak yapılan SDM iş akışına odaklanıldığından bazı ayrıntılı bilgiler atlanmıştır.
WorldClim V1 Bioclim: Bu veri kümesi, aylık sıcaklık ve yağış verilerinden elde edilen 19 biyoklimatik değişken sağlar. 1960-1991 dönemini kapsar ve 927,67 metre çözünürlüğe sahiptir.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
NASA SRTM Dijital Yükseklik 30 m: Bu veri kümesi, Uzay Mekiği Radar Topografya Misyonu'ndan (SRTM) elde edilen dijital yükseklik verilerini içerir. Veriler esas olarak 2000 yılı civarında toplanmış olup yaklaşık 30 metre (1 yay saniyesi) çözünürlükte sağlanır. Aşağıdaki kod, SRTM verilerinden yükseklik, eğim, yön ve tepe gölgesi katmanlarını hesaplar.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m: Landsat'ten alınan Vegetation Continuous Fields (VCF) veri kümesi, bitki yüksekliği 5 metreden fazla olduğunda dikey olarak yansıtılan bitki örtüsü oranını tahmin eder. Bu veri kümesi, 2000, 2005, 2010 ve 2015 yılları merkez alınarak 30 metre çözünürlükle dört zaman aralığı için sağlanır. Burada, bu dört zaman aralığındaki ortanca değerler kullanılır.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (Biyoklimatik değişkenler), terrain (topografya) ve median_tcc (ağaç örtüsü) tek bir çok bantlı görüntüde birleştirilir. elevation bant, terrain arasından seçilir ve elevation değerinin 0 değerinden büyük olduğu konumlar için bir watermask oluşturulur. Bu, deniz seviyesinin altındaki bölgeleri (ör. okyanus) maskeler ve araştırmacıyı, İB'deki çeşitli çevresel faktörleri kapsamlı bir şekilde analiz etmeye hazırlar.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
Yüksek oranda ilişkili kestirici değişkenler bir modele birlikte dahil edildiğinde çoklu bağlantı sorunları ortaya çıkabilir. Çoklu doğrusallık, bir modeldeki bağımsız değişkenler arasında güçlü doğrusal ilişkiler olduğunda ortaya çıkan bir olgudur ve modelin katsayılarının (ağırlıklarının) tahmininde kararsızlığa yol açar. Bu kararsızlık, modelin güvenilirliğini azaltabilir ve yeni verilerle ilgili tahmin veya yorum yapmayı zorlaştırabilir. Bu nedenle, çoklu doğrusallığı dikkate alacak ve tahmin değişkenlerini seçme sürecine devam edeceğiz.
Öncelikle 5.000 rastgele nokta oluşturacağız ve ardından bu noktalarda tek bir çok bantlı görüntünün tahmin değişkeni değerlerini çıkaracağız.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
Her nokta için çıkarılan tahminci değerlerini DataFrame'e dönüştürürüz ve ardından ilk satırı kontrol ederiz.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
Belirli tahmin değişkenleri arasındaki Spearman korelasyon katsayılarını hesaplama ve bunları ısı haritasında görselleştirme.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
Spearman korelasyon katsayısı, tahmin değişkenleri arasındaki genel ilişkileri anlamak için yararlıdır ancak birden çok değişkenin nasıl etkileşime girdiğini doğrudan değerlendirmez ve özellikle çoklu doğrusallığı tespit etmez.
Varyans enflasyon faktörü (VIF), çoklu doğrusallığı değerlendirmek ve değişken seçimini yönlendirmek için kullanılan istatistiksel bir metriktir. Her bir bağımsız değişkenin diğer bağımsız değişkenlerle doğrusal ilişki derecesini gösterir. Yüksek VIF değerleri, çoklu doğrusallığın kanıtı olabilir.
Genellikle VIF değerleri 5 veya 10'u aştığında değişkenin diğer değişkenlerle güçlü bir korelasyonu olduğu anlaşılır. Bu durum, modelin kararlılığını ve yorumlanabilirliğini tehlikeye atabilir. Bu eğitimde, değişken seçimi için 10'dan küçük VIF değerleri ölçütü kullanılmıştır. Aşağıdaki 6 değişken, VIF'ye göre seçildi.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
Ardından, seçilen 6 tahmin değişkenini haritada görselleştirelim.
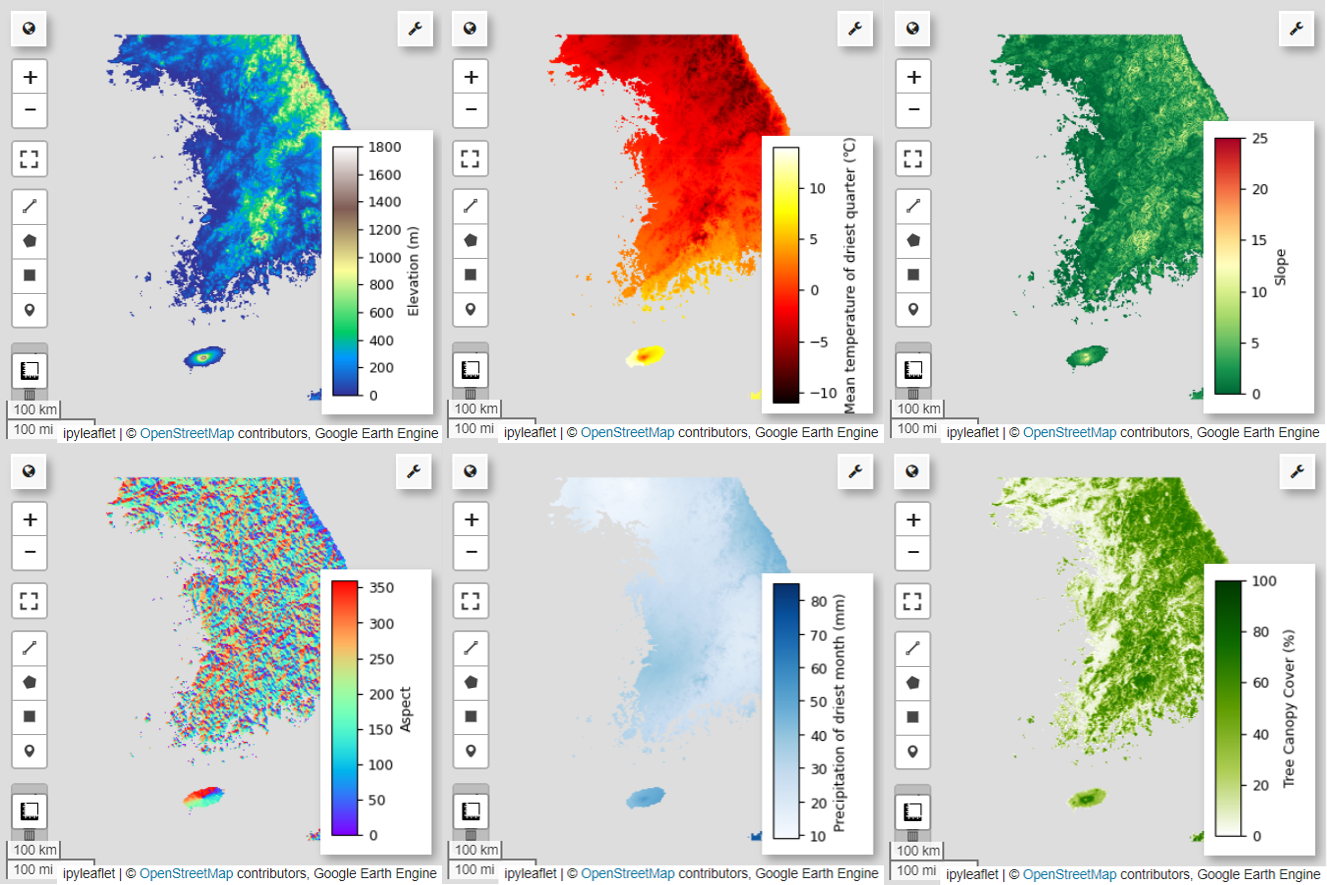
Harita görselleştirmesi için kullanılabilen paletleri aşağıdaki kodu kullanarak keşfedebilirsiniz. Örneğin, terrain paleti şu şekilde görünür.
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
Sözde yokluk verilerinin oluşturulması
SDM sürecinde, bir tür için giriş verilerinin seçimi temel olarak iki yöntem kullanılarak yapılır:
Varlık-Arka Plan Yöntemi: Bu yöntemde, belirli bir türün görüldüğü konumlar (varlık) türün görülmediği diğer konumlarla (arka plan) karşılaştırılır. Burada arka plan verileri, türün bulunmadığı alanları ifade etmez. Bunun yerine, çalışma alanının genel çevre koşullarını yansıtacak şekilde ayarlanır. Türlerin yaşayabileceği uygun ortamları daha az uygun olanlardan ayırt etmek için kullanılır.
Varlık-Yokluk Yöntemi: Bu yöntemde, türün gözlemlendiği (varlık) konumlar, kesin olarak gözlemlenmediği (yokluk) konumlarla karşılaştırılır. Burada, yokluk verileri türün bulunmadığı bilinen belirli konumları temsil eder. Çalışma alanının genel çevre koşullarını yansıtmaz, bunun yerine türün bulunmadığı tahmin edilen yerleri gösterir.
Uygulamada, gerçek yokluk verilerini toplamak genellikle zordur. Bu nedenle, yapay olarak oluşturulan sahte yokluk verileri sıklıkla kullanılır. Ancak, yapay olarak oluşturulan sözde yokluk noktaları gerçek yokluk alanlarını doğru şekilde yansıtmayabileceğinden bu yöntemin sınırlamalarını ve olası hatalarını kabul etmek önemlidir.
Bu iki yöntem arasındaki seçim; verilerin kullanılabilirliğine, araştırma hedeflerine, modelin doğruluğuna ve güvenilirliğine, ayrıca zamana ve kaynaklara bağlıdır. Burada, "Var-Yok" yöntemini kullanarak modelleme yapmak için GBIF'den toplanan oluşum verilerini ve yapay olarak oluşturulan sözde yokluk verilerini kullanacağız.
Sözde yokluk verilerinin oluşturulması "çevresel profilleme yaklaşımı" ile yapılır ve belirli adımlar şu şekildedir:
k-means kümeleme kullanılarak çevresel sınıflandırma: Çalışma alanındaki pikselleri iki kümeye ayırmak için Öklid mesafesine dayalı k-means kümeleme algoritması kullanılır. Bir küme, rastgele seçilen 100 varlık konumuna benzer çevresel özelliklere sahip alanları temsil ederken diğer küme, farklı özelliklere sahip alanları temsil eder.
Benzer Olmayan Kümelerde Sahte Yokluk Verileri Oluşturma: İlk adımda tanımlanan ikinci kümede (varlık verilerinden farklı çevresel özelliklere sahip) rastgele oluşturulmuş sahte yokluk noktaları oluşturulur. Bu sözde yokluk noktaları, türün bulunmasının beklenmediği konumları temsil eder.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
Model uyumu ve tahmin
Şimdi verileri eğitim verileri ve test verileri olarak ayıracağız. Eğitim verileri, modeli eğiterek en uygun parametreleri bulmak için kullanılırken test verileri, daha önce eğitilmiş modeli değerlendirmek için kullanılır. Bu bağlamda dikkate alınması gereken önemli bir kavram, mekansal otokorelasyondur.
Mekansal otokorelasyon, SDM'de Tobler'ın yasasıyla ilişkili temel bir unsurdur. "Her şey birbiriyle ilişkilidir ancak yakın şeyler uzak şeylerden daha çok ilişkilidir" kavramını yansıtır. Mekansal otokorelasyon, türlerin konumu ile çevresel değişkenler arasındaki önemli ilişkiyi ifade eder. Ancak eğitim ve test verileri arasında mekansal otokorelasyon varsa iki veri kümesi arasındaki bağımsızlık tehlikeye girebilir. Bu durum, modelin genelleme yeteneğinin değerlendirilmesini önemli ölçüde etkiler.
Bu sorunu çözmek için kullanılan yöntemlerden biri, verileri eğitim ve test veri kümelerine bölmeyi içeren mekansal blok çapraz doğrulama tekniğidir. Bu teknikte, mekansal otokorelasyonun etkisini azaltmak için veriler birden fazla bloğa bölünür ve her blok bağımsız olarak eğitim ve test veri kümeleri şeklinde kullanılır. Bu, veri kümeleri arasındaki bağımsızlığı artırarak modelin genelleme yeteneğinin daha doğru bir şekilde değerlendirilmesini sağlar.
İşlem şu şekildedir:
- Mekansal blokların oluşturulması: Veri kümesinin tamamını eşit boyutlu mekansal bloklara bölün (ör. 50x50 km).
- Eğitim ve test kümelerinin atanması: Her mekansal blok, eğitim kümesine (%70) veya test kümesine (%30) rastgele atanır. Bu, modelin belirli alanlardaki verilere aşırı uyum sağlamasını önler ve daha genelleştirilmiş sonuçlar elde etmeyi amaçlar.
- İterasyonlu çapraz doğrulama: Tüm süreç n kez tekrarlanır (ör. 10 kez). Her yinelemede bloklar, modelin kararlılığını ve güvenilirliğini artırmak amacıyla tekrar rastgele bir şekilde eğitim ve test kümelerine ayrılır.
- Sözde yokluk verileri oluşturma: Her yinelemede, modelin performansını değerlendirmek için rastgele sözde yokluk verileri oluşturulur.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
Şimdi modeli eğitebiliriz. Bir modeli uydurmak, verilerdeki kalıpları anlamayı ve modelin parametrelerini (ağırlıklar ve önyargılar) buna göre ayarlamayı içerir. Bu süreç, modele yeni veriler sunulduğunda daha doğru tahminler yapmasını sağlar. Bu amaçla, modele uyması için SDM() adlı bir işlev tanımladık.
Rastgele Orman algoritmasını kullanacağız.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
Mekansal bloklar sırasıyla% 70 model eğitimi ve% 30 model testi için ayrılır. Sözde yokluk verileri, her yinelemede her eğitim ve test kümesinde rastgele oluşturulur. Bu nedenle, her yürütme, model eğitimi ve testi için farklı varlık ve sözde yokluk verileri kümeleri üretir.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
Artık peri pitası için yaşam alanı uygunluk haritasını ve potansiyel dağılım haritasını görselleştirebiliriz. Bu durumda, yaşam alanı uygunluk haritası, tüm resimlerdeki her piksel konumunun ortalamasını hesaplamak için mean() işlevi kullanılarak oluşturulur. Potansiyel dağıtım haritası ise tüm resimlerdeki her piksel konumunda en sık görünen değeri belirlemek için mode() işlevi kullanılarak oluşturulur.
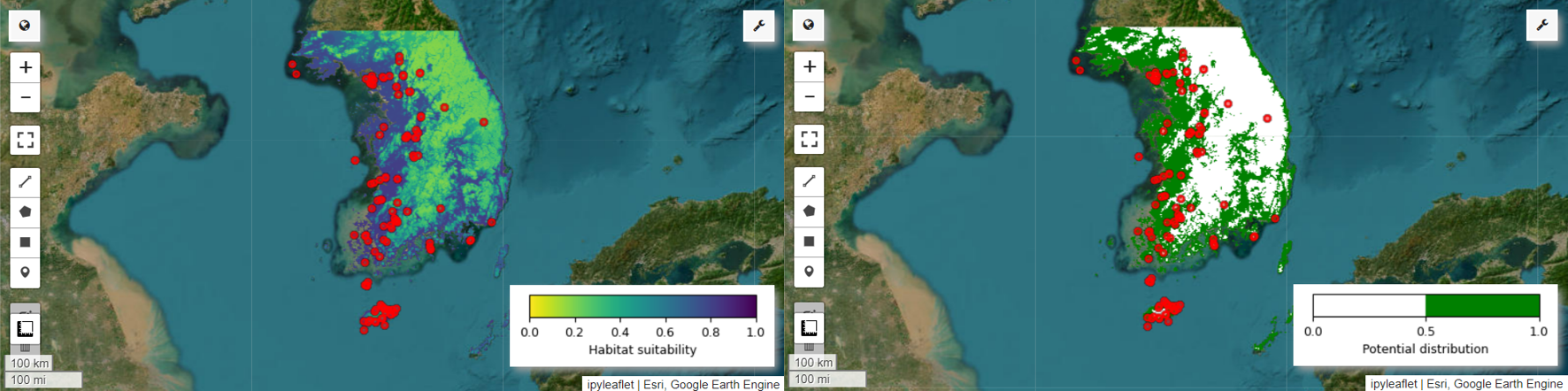
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
Değişken önemi ve doğruluk değerlendirmesi
Rastgele orman (ee.Classifier.smileRandomForest), tahminlerde bulunmak için birden fazla karar ağacı oluşturarak çalışan toplu öğrenme yöntemlerinden biridir. Her karar ağacı, verilerin farklı alt kümelerinden bağımsız olarak öğrenir ve sonuçları, daha doğru ve istikrarlı tahminler sağlamak için toplanır.
Değişken önemi, her bir değişkenin Rastgele Orman modelindeki tahminler üzerindeki etkisini değerlendiren bir ölçüdür. Ortalama değişken önemini hesaplamak ve yazdırmak için daha önce tanımlanmış importances_list değerini kullanacağız.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
Test veri kümelerini kullanarak her çalıştırma için AUC-ROC ve AUC-PR değerlerini hesaplarız. Ardından, n yineleme üzerinden ortalama AUC-ROC ve AUC-PR değerlerini hesaplarız.
AUC-ROC, eşik değiştikçe hassasiyet ile özgüllük arasındaki ilişkiyi gösteren "Hassasiyet (Geri Çağırma) ve 1-Özgüllük" grafiğinin eğrisinin altındaki alanı ifade eder. Özgüllük, gözlemlenen tüm gerçekleşmeme durumlarına dayanır. Bu nedenle, AUC-ROC, karmaşıklık matrisinin tüm çeyreklerini kapsar.
AUC-PR, eşik değiştikçe hassasiyet ile geri çağırma arasındaki ilişkiyi gösteren "Hassasiyet ve Geri Çağırma (Duyarlılık)" grafiğinin eğrisi altındaki alanı ifade eder. Hassasiyet, tahmin edilen tüm oluşumlara dayanır. Bu nedenle, AUC-PR gerçek negatifleri (TN) içermez.
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
Bu eğitimde, Tür Dağılımı Modellemesi (SDM) için Google Earth Engine'in (GEE) kullanımına dair pratik bir örnek verilmiştir. Önemli bir sonuç, GEE'nin SDM alanındaki çok yönlülüğü ve esnekliğidir. Earth Engine'in güçlü coğrafi veri işleme özelliklerinden yararlanmak, araştırmacılar ve doğa koruma uzmanları için gezegenimizdeki biyolojik çeşitliliği anlamak ve korumak adına sonsuz olanaklar sunuyor. Bu eğitimden edindikleri bilgi ve becerileri uygulayarak bireyler, ekolojik araştırmaların bu büyüleyici alanını keşfedebilir ve bu alana katkıda bulunabilir.
 Google Colab'de çalıştır
Google Colab'de çalıştır
 Kaynağı GitHub'da görüntüleyin
Kaynağı GitHub'da görüntüleyin