Dalam tutorial ini, metodologi Pemodelan Distribusi Spesies menggunakan Google Earth Engine akan diperkenalkan. Ringkasan singkat tentang Pemodelan Distribusi Spesies akan diberikan, diikuti dengan proses memprediksi dan menganalisis habitat spesies burung langka yang dikenal sebagai burung pitta peri (nama ilmiah: Pitta nympha).
Jalankan saya terlebih dahulu
Jalankan sel berikut untuk melakukan inisialisasi API. Output akan berisi petunjuk tentang cara memberikan akses notebook ini ke Earth Engine menggunakan akun Anda.
import ee
# Trigger the authentication flow.
ee.Authenticate()
# Initialize the library.
ee.Initialize(project='my-project')
Ringkasan singkat Pemodelan Distribusi Spesies
Mari kita pelajari apa itu model distribusi spesies, keuntungan menggunakan Google Earth Engine untuk pemrosesannya, data yang diperlukan untuk model, dan cara alur kerja disusun.
Apa yang dimaksud dengan Pemodelan Distribusi Spesies?
Pemodelan Distribusi Spesies (SDM di bawah) adalah metodologi paling umum yang digunakan untuk memperkirakan distribusi geografis aktual atau potensial suatu spesies. Hal ini melibatkan karakterisasi kondisi lingkungan yang sesuai untuk spesies tertentu, lalu mengidentifikasi lokasi distribusi kondisi yang sesuai ini secara geografis.
SDM telah muncul sebagai komponen penting dalam perencanaan konservasi dalam beberapa tahun terakhir, dan berbagai teknik pemodelan telah dikembangkan untuk tujuan ini. Penerapan SDM di Google Earth Engine (GEE di bawah) memberikan akses mudah ke data lingkungan skala besar, beserta kemampuan komputasi yang canggih dan dukungan untuk algoritma machine learning, sehingga memungkinkan pemodelan yang cepat.
Data yang Diperlukan untuk SDM
SDM biasanya memanfaatkan hubungan antara catatan kemunculan spesies yang diketahui dan variabel lingkungan untuk mengidentifikasi kondisi yang dapat menopang populasi. Dengan kata lain, dua jenis data input model diperlukan:
- Catatan kemunculan spesies yang diketahui
- Berbagai variabel lingkungan
Data ini dimasukkan ke dalam algoritma untuk mengidentifikasi kondisi lingkungan yang terkait dengan keberadaan spesies.
Alur kerja SDM menggunakan GEE
Alur kerja untuk SDM menggunakan GEE adalah sebagai berikut:
- Pengumpulan dan prapemrosesan data kemunculan spesies
- Definisi Area Minat
- Penambahan variabel lingkungan GEE
- Pembuatan data pseudo-absence
- Pencocokan dan prediksi model
- Penilaian akurasi dan kepentingan variabel
Prediksi dan Analisis Habitat Menggunakan GEE
Pitta peri (Pitta nympha) akan digunakan sebagai studi kasus untuk mendemonstrasikan penerapan SDM berbasis GEE. Meskipun spesies khusus ini telah dipilih untuk satu contoh, peneliti dapat menerapkan metodologi ini ke spesies target yang diminati dengan sedikit modifikasi pada kode sumber yang diberikan.
Burung pitta peri adalah burung migran musim panas dan burung migran yang lewat yang langka di Korea Selatan, yang area distribusinya meluas karena pemanasan iklim baru-baru ini di Semenanjung Korea. Hewan ini diklasifikasikan sebagai spesies langka, satwa liar terancam punah kelas II, Monumen Alam No. 204, dievaluasi sebagai Punah di Tingkat Regional (RE) dalam Daftar Merah Nasional, dan Rentan (VU) menurut kategori IUCN.
Melakukan SDM untuk perencanaan konservasi burung paok zamrud tampaknya cukup berharga. Sekarang, mari kita lanjutkan dengan prediksi dan analisis habitat melalui GEE.
Pertama, library Python diimpor.Pernyataan import memasukkan seluruh konten modul, sedangkan pernyataan from import memungkinkan impor objek tertentu dari modul.
# Import libraries
import geemap
import geemap.colormaps as cm
import pandas as pd, geopandas as gpd
import numpy as np, matplotlib.pyplot as plt
import os, requests, math, random
from ipyleaflet import TileLayer
from statsmodels.stats.outliers_influence import variance_inflation_factor
Pengumpulan dan Pemrosesan Awal Data Keberadaan Spesies
Sekarang, mari kita kumpulkan data kemunculan untuk burung pitta peri. Meskipun saat ini Anda tidak memiliki akses ke data kemunculan untuk spesies yang diminati, Anda dapat memperoleh data observasi tentang spesies tertentu melalui GBIF API. GBIF API adalah antarmuka yang memungkinkan akses ke data distribusi spesies yang disediakan oleh GBIF, sehingga pengguna dapat menelusuri, memfilter, dan mendownload data, serta mendapatkan berbagai informasi terkait spesies.
Dalam kode di bawah, variabel species_name diberi nama ilmiah spesies (misalnya, Pitta nympha untuk Fairy pitta), dan variabel country_code diberi kode negara (misalnya, KR untuk Korea Selatan). Variabel base_url menyimpan alamat GBIF API. params adalah kamus yang berisi parameter yang akan digunakan dalam permintaan API:
scientificName: Menetapkan nama ilmiah spesies yang akan dicari.country: Membatasi penelusuran ke negara tertentu.hasCoordinate: Memastikan hanya data dengan koordinat (benar) yang ditelusuri.basisOfRecord: Hanya memilih catatan pengamatan manusia (HUMAN_OBSERVATION).limit: Menetapkan jumlah maksimum hasil yang ditampilkan menjadi 10000.
def get_gbif_species_data(species_name, country_code):
"""
Retrieves observational data for a specific species using the GBIF API and returns it as a pandas DataFrame.
Parameters:
species_name (str): The scientific name of the species to query.
country_code (str): The country code of the where the observation data will be queried.
Returns:
pd.DataFrame: A pandas DataFrame containing the observational data.
"""
base_url = "https://api.gbif.org/v1/occurrence/search"
params = {
"scientificName": species_name,
"country": country_code,
"hasCoordinate": "true",
"basisOfRecord": "HUMAN_OBSERVATION",
"limit": 10000,
}
try:
response = requests.get(base_url, params=params)
response.raise_for_status() # Raises an exception for a response error.
data = response.json()
occurrences = data.get("results", [])
if occurrences: # If data is present
df = pd.json_normalize(occurrences)
return df
else:
print("No data found for the given species and country code.")
return pd.DataFrame() # Returns an empty DataFrame
except requests.RequestException as e:
print(f"Request failed: {e}")
return pd.DataFrame() # Returns an empty DataFrame in case of an exception
Dengan menggunakan parameter yang ditetapkan sebelumnya, kita membuat kueri GBIF API untuk mendapatkan data pengamatan burung pitta peri (Pitta nympha), dan memuat hasilnya ke dalam DataFrame untuk memeriksa baris pertama. DataFrame adalah struktur data untuk menangani data berformat tabel, yang terdiri dari baris dan kolom. Jika perlu, DataFrame dapat disimpan sebagai file CSV dan dibaca kembali.
# Retrieve Fairy Pitta data
df = get_gbif_species_data("Pitta nympha", "KR")
"""
# Save DataFrame to CSV and read back in.
df.to_csv("pitta_nympha_data.csv", index=False)
df = pd.read_csv("pitta_nympha_data.csv")
"""
df.head(1) # Display the first row of the DataFrame
Selanjutnya, kita mengonversi DataFrame menjadi GeoDataFrame yang menyertakan kolom untuk informasi geografis (geometry) dan memeriksa baris pertama. GeoDataFrame dapat disimpan sebagai file GeoPackage (*.gpkg) dan dibaca kembali.
# Convert DataFrame to GeoDataFrame
gdf = gpd.GeoDataFrame(
df,
geometry=gpd.points_from_xy(df.decimalLongitude,
df.decimalLatitude),
crs="EPSG:4326"
)[["species", "year", "month", "geometry"]]
"""
# Convert GeoDataFrame to GeoPackage (requires pycrs module)
%pip install -U -q pycrs
gdf.to_file("pitta_nympha_data.gpkg", driver="GPKG")
gdf = gpd.read_file("pitta_nympha_data.gpkg")
"""
gdf.head(1) # Display the first row of the GeoDataFrame
Kali ini, kita telah membuat fungsi untuk memvisualisasikan distribusi data menurut tahun dan bulan dari GeoDataFrame dan menampilkannya sebagai grafik, yang kemudian dapat disimpan sebagai file gambar. Penggunaan peta panas memungkinkan kita memahami dengan cepat frekuensi kemunculan spesies menurut tahun dan bulan, sehingga memberikan visualisasi intuitif tentang perubahan dan pola temporal dalam data. Hal ini memungkinkan identifikasi pola temporal dan variasi musiman dalam data kemunculan spesies, serta deteksi cepat pencilan atau masalah kualitas dalam data.
# Yearly and monthly data distribution heatmap
def plot_heatmap(gdf, h_size=8):
statistics = gdf.groupby(["month", "year"]).size().unstack(fill_value=0)
# Heatmap
plt.figure(figsize=(h_size, h_size - 6))
heatmap = plt.imshow(
statistics.values, cmap="YlOrBr", origin="upper", aspect="auto"
)
# Display values above each pixel
for i in range(len(statistics.index)):
for j in range(len(statistics.columns)):
plt.text(
j, i, statistics.values[i, j], ha="center", va="center", color="black"
)
plt.colorbar(heatmap, label="Count")
plt.title("Monthly Species Count by Year")
plt.xlabel("Year")
plt.ylabel("Month")
plt.xticks(range(len(statistics.columns)), statistics.columns)
plt.yticks(range(len(statistics.index)), statistics.index)
plt.tight_layout()
plt.savefig("heatmap_plot.png")
plt.show()
plot_heatmap(gdf)
Data dari tahun 1995 sangat jarang, dengan kesenjangan yang signifikan dibandingkan dengan tahun-tahun lainnya, dan bulan Agustus serta September juga memiliki sampel terbatas dan menunjukkan karakteristik musiman yang berbeda dibandingkan dengan periode lainnya. Mengecualikan data ini dapat membantu meningkatkan stabilitas dan kemampuan prediktif model.
Namun, penting untuk diperhatikan bahwa mengecualikan data dapat meningkatkan kemampuan generalisasi model, tetapi juga dapat menyebabkan hilangnya informasi berharga yang relevan dengan tujuan penelitian. Oleh karena itu, keputusan tersebut harus dibuat dengan pertimbangan yang cermat.
# Filtering data by year and month
filtered_gdf = gdf[
(~gdf['year'].eq(1995)) &
(~gdf['month'].between(8, 9))
]
Sekarang, GeoDataFrame yang difilter dikonversi menjadi objek Google Earth Engine.
# Convert GeoDataFrame to Earth Engine object
data_raw = geemap.geopandas_to_ee(filtered_gdf)
Selanjutnya, kita akan menentukan ukuran piksel raster hasil SDM sebagai resolusi 1 km.
# Spatial resolution setting (meters)
grain_size = 1000
Jika beberapa titik kejadian ada dalam piksel raster resolusi 1 km yang sama, kemungkinan besar titik tersebut memiliki kondisi lingkungan yang sama di lokasi geografis yang sama. Penggunaan data tersebut secara langsung dalam analisis dapat menimbulkan bias pada hasilnya.
Dengan kata lain, kita perlu membatasi potensi dampak bias pengambilan sampel geografis. Untuk mencapainya, kita hanya akan mempertahankan satu lokasi dalam setiap piksel 1 km dan menghapus semua lokasi lainnya, sehingga model dapat mencerminkan kondisi lingkungan secara lebih objektif.
def remove_duplicates(data, grain_size):
# Select one occurrence record per pixel at the chosen spatial resolution
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
rand_point_vals = random_raster.sampleRegions(
collection=ee.FeatureCollection(data), geometries=True
)
return rand_point_vals.distinct("random")
data = remove_duplicates(data_raw, grain_size)
# Before selection and after selection
print("Original data size:", data_raw.size().getInfo())
print("Final data size:", data.size().getInfo())
Visualisasi yang membandingkan bias pengambilan sampel geografis sebelum prapemrosesan (berwarna biru) dan setelah prapemrosesan (berwarna merah) ditampilkan di bawah. Untuk memfasilitasi perbandingan, peta telah dipusatkan pada area dengan konsentrasi tinggi koordinat kemunculan burung Paok zamrud di Taman Nasional Hallasan.
# Visualization of geographic sampling bias before (blue) and after (red) preprocessing
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
# Add the random raster layer
random_raster = ee.Image.random().reproject("EPSG:4326", None, grain_size)
Map.addLayer(
random_raster,
{"min": 0, "max": 1, "palette": ["black", "white"], "opacity": 0.5},
"Random Raster",
)
# Add the original data layer in blue
Map.addLayer(data_raw, {"color": "blue"}, "Original data")
# Add the final data layer in red
Map.addLayer(data, {"color": "red"}, "Final data")
# Set the center of the map to the coordinates
Map.setCenter(126.712, 33.516, 14)
Map
Definisi Area Minat
Menentukan Area Minat (AOI di bawah) mengacu pada istilah yang digunakan oleh peneliti untuk menunjukkan area geografis yang ingin mereka analisis. Maknanya mirip dengan istilah Area Studi.
Dalam konteks ini, kami mendapatkan kotak pembatas geometri lapisan titik kejadian dan membuat buffer 50 kilometer di sekitarnya (dengan toleransi maksimum 1.000 meter) untuk menentukan AOI.
# Define the AOI
aoi = data.geometry().bounds().buffer(distance=50000, maxError=1000)
# Add the AOI to the map
outline = ee.Image().byte().paint(
featureCollection=aoi, color=1, width=3)
Map.remove_layer("Random Raster")
Map.addLayer(outline, {'palette': 'FF0000'}, "AOI")
Map.centerObject(aoi, 6)
Map
Penambahan variabel lingkungan GEE
Sekarang, tambahkan variabel lingkungan ke analisis. GEE menyediakan berbagai set data untuk variabel lingkungan seperti suhu, presipitasi, elevasi, penutupan lahan, dan medan. Set data ini memungkinkan kami menganalisis secara komprehensif berbagai faktor yang dapat memengaruhi preferensi habitat burung Paok Lawe.
Pemilihan variabel lingkungan GEE dalam SDM harus mencerminkan karakteristik preferensi habitat spesies. Untuk melakukannya, penelitian sebelumnya dan tinjauan literatur tentang preferensi habitat burung Paok zamrud harus dilakukan. Tutorial ini terutama berfokus pada alur kerja SDM menggunakan GEE, sehingga beberapa detail mendalam tidak disertakan.
WorldClim V1 Bioclim: Set data ini menyediakan 19 variabel bioklimatik yang berasal dari data suhu dan curah hujan bulanan. Dataset ini mencakup periode dari tahun 1960 hingga 1991 dan memiliki resolusi 927,67 meter.
# WorldClim V1 Bioclim
bio = ee.Image("WORLDCLIM/V1/BIO")
NASA SRTM Digital Elevation 30m: Set data ini berisi data elevasi digital dari Shuttle Radar Topography Mission (SRTM). Data ini terutama dikumpulkan sekitar tahun 2000 dan disediakan pada resolusi sekitar 30 meter (1 detik busur). Kode berikut menghitung lapisan elevasi, kemiringan, aspek, dan hillshade dari data SRTM.
# NASA SRTM Digital Elevation 30m
terrain = ee.Algorithms.Terrain(ee.Image("USGS/SRTMGL1_003"))
Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m: Set data Vegetation Continuous Fields (VCF) dari Landsat memperkirakan proporsi tutupan vegetasi yang diproyeksikan secara vertikal jika tinggi vegetasi lebih dari 5 meter. Set data ini disediakan untuk empat periode waktu yang berpusat di sekitar tahun 2000, 2005, 2010, dan 2015, dengan resolusi 30 meter. Di sini, nilai median dari empat jangka waktu ini digunakan.
# Global Forest Cover Change (GFCC) Tree Cover Multi-Year Global 30m
tcc = ee.ImageCollection("NASA/MEASURES/GFCC/TC/v3")
median_tcc = (
tcc.filterDate("2000-01-01", "2015-12-31")
.select(["tree_canopy_cover"], ["TCC"])
.median()
)
bio (Variabel bioklimatik), terrain (topografi), dan median_tcc (cakupan tajuk pohon) digabungkan menjadi satu gambar multiband. Rentang elevation dipilih dari terrain, dan watermask dibuat untuk lokasi tempat elevation lebih besar dari 0. Hal ini menutupi wilayah di bawah permukaan laut (misalnya, lautan) dan mempersiapkan peneliti untuk menganalisis berbagai faktor lingkungan untuk AOI secara komprehensif.
# Combine bands into a multi-band image
predictors = bio.addBands(terrain).addBands(median_tcc)
# Create a water mask
watermask = terrain.select('elevation').gt(0)
# Mask out ocean pixels and clip to the area of interest
predictors = predictors.updateMask(watermask).clip(aoi)
Jika variabel prediktor yang sangat berkorelasi disertakan bersama dalam model, masalah multikolinearitas dapat muncul. Multikolinearitas adalah fenomena yang terjadi ketika ada hubungan linear yang kuat di antara variabel independen dalam model, sehingga menyebabkan ketidakstabilan dalam estimasi koefisien (bobot) model. Ketidakstabilan ini dapat mengurangi keandalan model dan membuat prediksi atau interpretasi untuk data baru menjadi sulit. Oleh karena itu, kita akan mempertimbangkan multikolinearitas dan melanjutkan proses pemilihan variabel prediktor.
Pertama, kita akan membuat 5.000 titik acak,lalu mengekstrak nilai variabel prediktor dari satu gambar multiband di titik-titik tersebut.
# Generate 5,000 random points
data_cor = predictors.sample(scale=grain_size, numPixels=5000, geometries=True)
# Extract predictor variable values
pvals = predictors.sampleRegions(collection=data_cor, scale=grain_size)
Kita akan mengonversi nilai prediktor yang diekstrak untuk setiap titik menjadi DataFrame, lalu memeriksa baris pertama.
# Converting predictor values from Earth Engine to a DataFrame
pvals_df = geemap.ee_to_df(pvals)
pvals_df.head(1)
# Displaying the columns
columns = pvals_df.columns
print(columns)
Menghitung koefisien korelasi Spearman antara variabel prediktor yang diberikan dan memvisualisasikannya dalam peta panas.
def plot_correlation_heatmap(dataframe, h_size=10, show_labels=False):
# Calculate Spearman correlation coefficients
correlation_matrix = dataframe.corr(method="spearman")
# Create a heatmap
plt.figure(figsize=(h_size, h_size-2))
plt.imshow(correlation_matrix, cmap='coolwarm', interpolation='nearest')
# Optionally display values on the heatmap
if show_labels:
for i in range(correlation_matrix.shape[0]):
for j in range(correlation_matrix.shape[1]):
plt.text(j, i, f"{correlation_matrix.iloc[i, j]:.2f}",
ha='center', va='center', color='white', fontsize=8)
columns = dataframe.columns.tolist()
plt.xticks(range(len(columns)), columns, rotation=90)
plt.yticks(range(len(columns)), columns)
plt.title("Variables Correlation Matrix")
plt.colorbar(label="Spearman Correlation")
plt.savefig('correlation_heatmap_plot.png')
plt.show()
# Plot the correlation heatmap of variables
plot_correlation_heatmap(pvals_df)
Koefisien korelasi Spearman berguna untuk memahami hubungan umum di antara variabel prediktor, tetapi tidak secara langsung menilai cara beberapa variabel berinteraksi, khususnya mendeteksi multikolinearitas.
Variance Inflation Factor (VIF di bawah) adalah metrik statistik yang digunakan untuk mengevaluasi multikolinearitas dan memandu pemilihan variabel. VIF menunjukkan tingkat hubungan linear setiap variabel independen dengan variabel independen lainnya, dan nilai VIF yang tinggi dapat menjadi bukti multikolinearitas.
Biasanya, jika nilai VIF melebihi 5 atau 10, hal ini menunjukkan bahwa variabel memiliki korelasi yang kuat dengan variabel lain, sehingga berpotensi mengganggu stabilitas dan kemampuan interpretasi model. Dalam tutorial ini, kriteria nilai VIF kurang dari 10 digunakan untuk pemilihan variabel. 6 variabel berikut dipilih berdasarkan VIF.
# Filter variables based on Variance Inflation Factor (VIF)
def filter_variables_by_vif(dataframe, threshold=10):
original_columns = dataframe.columns.tolist()
remaining_columns = original_columns[:]
while True:
vif_data = dataframe[remaining_columns]
vif_values = [
variance_inflation_factor(vif_data.values, i)
for i in range(vif_data.shape[1])
]
max_vif_index = vif_values.index(max(vif_values))
max_vif = max(vif_values)
if max_vif < threshold:
break
print(f"Removing '{remaining_columns[max_vif_index]}' with VIF {max_vif:.2f}")
del remaining_columns[max_vif_index]
filtered_data = dataframe[remaining_columns]
bands = filtered_data.columns.tolist()
print("Bands:", bands)
return filtered_data, bands
filtered_pvals_df, bands = filter_variables_by_vif(pvals_df)
# Variable Selection Based on VIF
predictors = predictors.select(bands)
# Plot the correlation heatmap of variables
plot_correlation_heatmap(filtered_pvals_df, h_size=6, show_labels=True)
Selanjutnya, mari kita visualisasikan 6 variabel prediktor yang dipilih di peta.
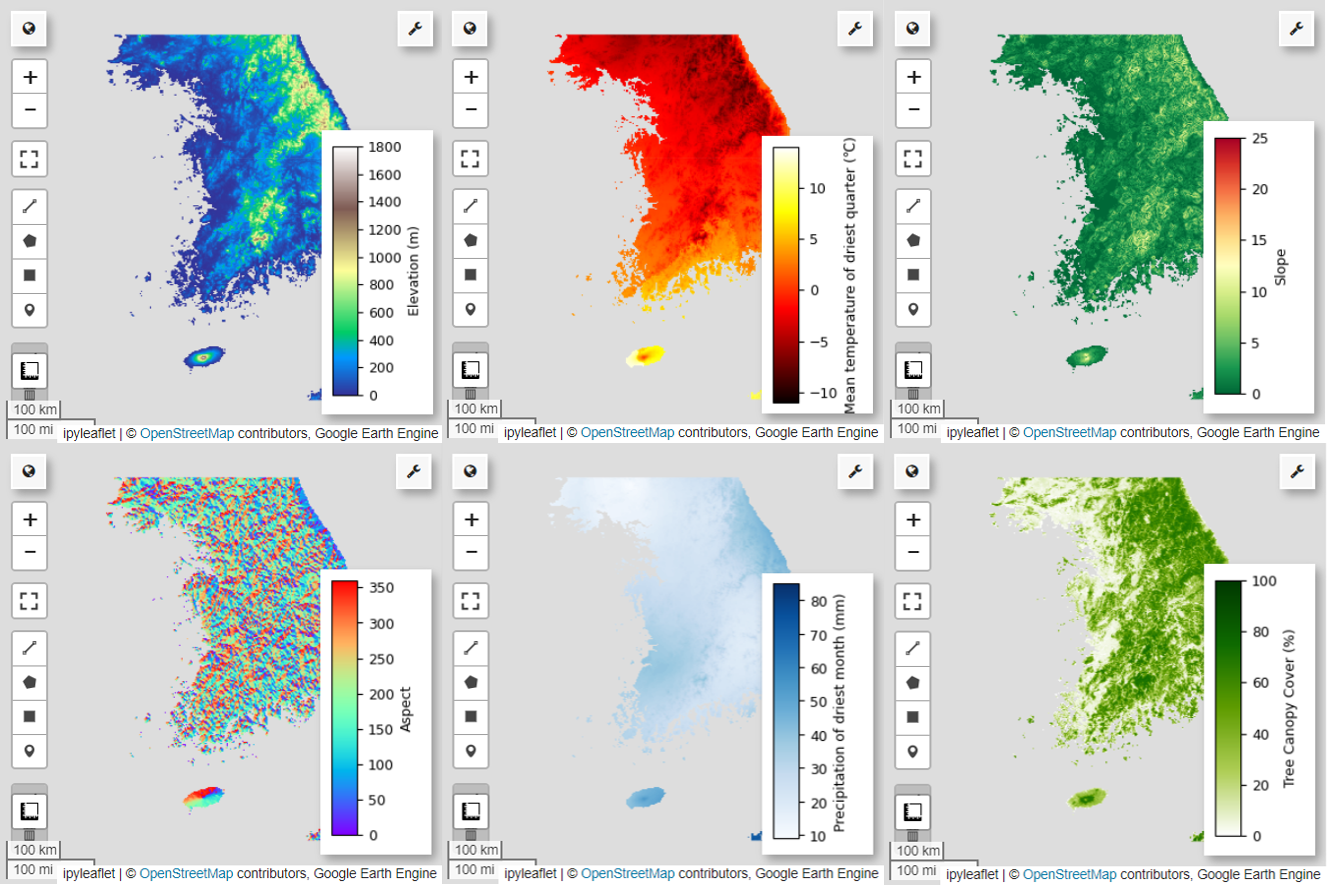
Anda dapat menjelajahi palet yang tersedia untuk visualisasi peta menggunakan kode berikut. Misalnya, palet terrain terlihat seperti ini.
cm.plot_colormaps(width=8.0, height=0.2)
cm.plot_colormap('terrain', width=8.0, height=0.2, orientation='horizontal')
# Elevation layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['elevation'], 'min': 0, 'max': 1800, 'palette': cm.palettes.terrain}
Map.addLayer(predictors, vis_params, 'elevation')
Map.add_colorbar(vis_params, label="Elevation (m)", orientation="vertical", layer_name="elevation")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio09
min_max_val = (
predictors.select("bio09")
.multiply(0.1)
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio09 (Mean temperature of driest quarter) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"min": math.floor(min_max_val["bio09_min"]),
"max": math.ceil(min_max_val["bio09_max"]),
"palette": cm.palettes.hot,
}
Map.addLayer(predictors.select("bio09").multiply(0.1), vis_params, "bio09")
Map.add_colorbar(
vis_params,
label="Mean temperature of driest quarter (℃)",
orientation="vertical",
layer_name="bio09",
)
Map.centerObject(aoi, 6)
Map
# Slope layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['slope'], 'min': 0, 'max': 25, 'palette': cm.palettes.RdYlGn_r}
Map.addLayer(predictors, vis_params, 'slope')
Map.add_colorbar(vis_params, label="Slope", orientation="vertical", layer_name="slope")
Map.centerObject(aoi, 6)
Map
# Aspect layer
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
vis_params = {'bands':['aspect'], 'min': 0, 'max': 360, 'palette': cm.palettes.rainbow}
Map.addLayer(predictors, vis_params, 'aspect')
Map.add_colorbar(vis_params, label="Aspect", orientation="vertical", layer_name="aspect")
Map.centerObject(aoi, 6)
Map
# Calculate the minimum and maximum values for bio14
min_max_val = (
predictors.select("bio14")
.reduceRegion(reducer=ee.Reducer.minMax(), scale=1000)
.getInfo()
)
# bio14 (Precipitation of driest month) layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["bio14"],
"min": math.floor(min_max_val["bio14_min"]),
"max": math.ceil(min_max_val["bio14_max"]),
"palette": cm.palettes.Blues,
}
Map.addLayer(predictors, vis_params, "bio14")
Map.add_colorbar(
vis_params,
label="Precipitation of driest month (mm)",
orientation="vertical",
layer_name="bio14",
)
Map.centerObject(aoi, 6)
Map
# TCC layer
Map = geemap.Map(layout={"height": "400px", "width": "800px"})
vis_params = {
"bands": ["TCC"],
"min": 0,
"max": 100,
"palette": ["ffffff", "afce56", "5f9c00", "0e6a00", "003800"],
}
Map.addLayer(predictors, vis_params, "TCC")
Map.add_colorbar(
vis_params, label="Tree Canopy Cover (%)", orientation="vertical", layer_name="TCC"
)
Map.centerObject(aoi, 6)
Map
Pembuatan data pseudo-absence
Dalam proses SDM, pemilihan data input untuk suatu spesies terutama dilakukan menggunakan dua metode:
Metode Kehadiran-Latar Belakang: Metode ini membandingkan lokasi tempat suatu spesies tertentu telah diamati (kehadiran) dengan lokasi lain tempat spesies tersebut tidak diamati (latar belakang). Di sini, data latar belakang tidak selalu berarti area tempat spesies tidak ada, tetapi disiapkan untuk mencerminkan kondisi lingkungan keseluruhan area studi. Hal ini digunakan untuk membedakan lingkungan yang cocok untuk keberadaan spesies tersebut dengan lingkungan yang kurang cocok.
Metode Kehadiran-Ketidakhadiran: Metode ini membandingkan lokasi tempat spesies telah diamati (kehadiran) dengan lokasi tempat spesies tersebut jelas tidak diamati (ketidakhadiran). Di sini, data tidak ada mewakili lokasi spesifik tempat spesies diketahui tidak ada. Hal ini tidak mencerminkan kondisi lingkungan secara keseluruhan di area studi, tetapi menunjukkan lokasi yang diperkirakan tidak dihuni oleh spesies tersebut.
Dalam praktiknya, sering kali sulit untuk mengumpulkan data ketidakhadiran yang sebenarnya, sehingga data pseudo-ketidakhadiran yang dibuat secara buatan sering digunakan. Namun, penting untuk mengakui keterbatasan dan potensi kesalahan metode ini, karena titik pseudo-absence yang dibuat secara artifisial mungkin tidak secara akurat mencerminkan area absence yang sebenarnya.
Pilihan antara kedua metode ini bergantung pada ketersediaan data, tujuan penelitian, akurasi dan keandalan model, serta waktu dan sumber daya. Di sini, kita akan menggunakan data kemunculan yang dikumpulkan dari GBIF dan data pseudo-ketidakhadiran yang dibuat secara buatan untuk membuat model menggunakan metode "Kehadiran-Ketidakhadiran".
Pembuatan data pseudo-absence akan dilakukan melalui "pendekatan pembuatan profil lingkungan", dan langkah-langkah spesifiknya adalah sebagai berikut:
Klasifikasi Lingkungan Menggunakan Pengelompokan k-means: Algoritma pengelompokan k-means, berdasarkan jarak Euclidean, akan digunakan untuk membagi piksel dalam area studi menjadi dua cluster. Satu cluster akan merepresentasikan area dengan karakteristik lingkungan yang serupa dengan 100 lokasi kehadiran yang dipilih secara acak, sedangkan cluster lainnya akan merepresentasikan area dengan karakteristik yang berbeda.
Pembuatan Data Pseudo-Absence dalam Kelompok yang Tidak Serupa: Dalam kelompok kedua yang diidentifikasi pada langkah pertama (yang memiliki karakteristik lingkungan yang berbeda dari data kehadiran), titik pseudo-absence yang dibuat secara acak akan dibuat. Titik pseudo-ketidakhadiran ini akan merepresentasikan lokasi tempat spesies tidak diperkirakan ada.
# Randomly select 100 locations for occurrence
pvals = predictors.sampleRegions(
collection=data.randomColumn().sort('random').limit(100),
properties=[],
scale=grain_size
)
# Perform k-means clustering
clusterer = ee.Clusterer.wekaKMeans(
nClusters=2,
distanceFunction="Euclidean"
).train(pvals)
cl_result = predictors.cluster(clusterer)
# Get cluster ID for locations similar to occurrence
cl_id = cl_result.sampleRegions(
collection=data.randomColumn().sort('random').limit(200),
properties=[],
scale=grain_size
)
# Define non-occurrence areas in dissimilar clusters
cl_id = ee.FeatureCollection(cl_id).reduceColumns(ee.Reducer.mode(),['cluster'])
cl_id = ee.Number(cl_id.get('mode')).subtract(1).abs()
cl_mask = cl_result.select(['cluster']).eq(cl_id)
# Presence location mask
presence_mask = data.reduceToImage(properties=['random'],
reducer=ee.Reducer.first()
).reproject('EPSG:4326', None,
grain_size).mask().neq(1).selfMask()
# Masking presence locations in non-occurrence areas and clipping to AOI
area_for_pa = presence_mask.updateMask(cl_mask).clip(aoi)
# Area for Pseudo-absence
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(area_for_pa, {'palette': 'black'}, 'AreaForPA')
Map.centerObject(aoi, 6)
Map
Pencocokan dan prediksi model
Sekarang kita akan membagi data menjadi data pelatihan dan data pengujian. Data pelatihan akan digunakan untuk menemukan parameter optimal dengan melatih model, sedangkan data pengujian akan digunakan untuk mengevaluasi model yang dilatih sebelumnya. Konsep penting yang perlu dipertimbangkan dalam konteks ini adalah autokorelasi spasial.
Autokorelasi spasial adalah elemen penting dalam SDM, yang terkait dengan hukum Tobler. Konsep ini mencakup gagasan bahwa "semua hal saling berhubungan, tetapi hal-hal yang berdekatan lebih berhubungan daripada hal-hal yang berjauhan". Autokorelasi spasial menunjukkan hubungan yang signifikan antara lokasi spesies dan variabel lingkungan. Namun, jika ada autokorelasi spasial antara data pelatihan dan pengujian, independensi antara kedua set data dapat terganggu. Hal ini sangat memengaruhi evaluasi kemampuan generalisasi model.
Salah satu metode untuk mengatasi masalah ini adalah teknik validasi silang blok spasial, yang melibatkan pembagian data menjadi set data pelatihan dan pengujian. Teknik ini melibatkan pembagian data menjadi beberapa blok, dengan setiap blok digunakan secara independen sebagai set data pelatihan dan pengujian untuk mengurangi dampak autokorelasi spasial. Hal ini meningkatkan independensi antar-kumpulan data, sehingga memungkinkan evaluasi yang lebih akurat terhadap kemampuan generalisasi model.
Prosedur spesifiknya adalah sebagai berikut:
- Pembuatan blok spasial: Membagi seluruh set data menjadi blok spasial berukuran sama (misalnya, 50x50 km).
- Penetapan set pelatihan dan pengujian: Setiap blok spasial ditetapkan secara acak ke set pelatihan (70%) atau set pengujian (30%). Hal ini mencegah model melakukan overfitting pada data dari area tertentu dan bertujuan untuk mencapai hasil yang lebih umum.
- Validasi silang berulang: Seluruh proses diulang sebanyak n kali (misalnya, 10 kali). Di setiap iterasi, blok dibagi lagi secara acak ke dalam set pelatihan dan pengujian, yang dimaksudkan untuk meningkatkan stabilitas dan keandalan model.
- Pembuatan data pseudo-absence: Pada setiap iterasi, data pseudo-absence dibuat secara acak untuk mengevaluasi performa model.
Scale = 50000
grid = watermask.reduceRegions(
collection=aoi.coveringGrid(scale=Scale, proj='EPSG:4326'),
reducer=ee.Reducer.mean()).filter(ee.Filter.neq('mean', None))
Map = geemap.Map(layout={'height':'400px', 'width':'800px'})
Map.addLayer(grid, {}, "Grid for spatial block cross validation")
Map.addLayer(outline, {'palette': 'FF0000'}, "Study Area")
Map.centerObject(aoi, 6)
Map
Sekarang kita dapat menyesuaikan model. Mencocokkan model melibatkan pemahaman pola dalam data dan menyesuaikan parameter model (bobot dan bias) yang sesuai. Proses ini memungkinkan model membuat prediksi yang lebih akurat saat disajikan dengan data baru. Untuk tujuan ini, kita telah menentukan fungsi yang disebut SDM() untuk menyesuaikan model.
Kita akan menggunakan algoritma Random Forest.
def sdm(x):
seed = ee.Number(x)
# Random block division for training and validation
rand_blk = ee.FeatureCollection(grid).randomColumn(seed=seed).sort("random")
training_grid = rand_blk.filter(ee.Filter.lt("random", split)) # Grid for training
testing_grid = rand_blk.filter(ee.Filter.gte("random", split)) # Grid for testing
# Presence points
presence_points = ee.FeatureCollection(data)
presence_points = presence_points.map(lambda feature: feature.set("PresAbs", 1))
tr_presence_points = presence_points.filter(
ee.Filter.bounds(training_grid)
) # Presence points for training
te_presence_points = presence_points.filter(
ee.Filter.bounds(testing_grid)
) # Presence points for testing
# Pseudo-absence points for training
tr_pseudo_abs_points = area_for_pa.sample(
region=training_grid,
scale=grain_size,
numPixels=tr_presence_points.size().add(300),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for training
tr_pseudo_abs_points = (
tr_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(tr_presence_points.size()))
)
tr_pseudo_abs_points = tr_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
te_pseudo_abs_points = area_for_pa.sample(
region=testing_grid,
scale=grain_size,
numPixels=te_presence_points.size().add(100),
seed=seed,
geometries=True,
)
# Same number of pseudo-absence points as presence points for testing
te_pseudo_abs_points = (
te_pseudo_abs_points.randomColumn()
.sort("random")
.limit(ee.Number(te_presence_points.size()))
)
te_pseudo_abs_points = te_pseudo_abs_points.map(lambda feature: feature.set("PresAbs", 0))
# Merge training and pseudo-absence points
training_partition = tr_presence_points.merge(tr_pseudo_abs_points)
testing_partition = te_presence_points.merge(te_pseudo_abs_points)
# Extract predictor variable values at training points
train_pvals = predictors.sampleRegions(
collection=training_partition,
properties=["PresAbs"],
scale=grain_size,
geometries=True,
)
# Random Forest classifier
classifier = ee.Classifier.smileRandomForest(
numberOfTrees=500,
variablesPerSplit=None,
minLeafPopulation=10,
bagFraction=0.5,
maxNodes=None,
seed=seed,
)
# Presence probability: Habitat suitability map
classifier_pr = classifier.setOutputMode("PROBABILITY").train(
train_pvals, "PresAbs", bands
)
classified_img_pr = predictors.select(bands).classify(classifier_pr)
# Binary presence/absence map: Potential distribution map
classifier_bin = classifier.setOutputMode("CLASSIFICATION").train(
train_pvals, "PresAbs", bands
)
classified_img_bin = predictors.select(bands).classify(classifier_bin)
return [
classified_img_pr,
classified_img_bin,
training_partition,
testing_partition,
], classifier_pr
Blok spasial dibagi menjadi 70% untuk pelatihan model dan 30% untuk pengujian model. Data pseudo-ketidakhadiran dibuat secara acak dalam setiap set pelatihan dan pengujian di setiap iterasi. Akibatnya, setiap eksekusi menghasilkan kumpulan data kehadiran dan pseudo-ketidakhadiran yang berbeda untuk pelatihan dan pengujian model.
split = 0.7
numiter = 10
# Random Seed
runif = lambda length: [random.randint(1, 1000) for _ in range(length)]
items = runif(numiter)
# Fixed seed
# items = [287, 288, 553, 226, 151, 255, 902, 267, 419, 538]
results_list = [] # Initialize SDM results list
importances_list = [] # Initialize variable importance list
for item in items:
result, trained = sdm(item)
# Accumulate SDM results into the list
results_list.extend(result)
# Accumulate variable importance into the list
importance = ee.Dictionary(trained.explain()).get('importance')
importances_list.extend(importance.getInfo().items())
# Flatten the SDM results list
results = ee.List(results_list).flatten()
Sekarang kita dapat memvisualisasikan peta kesesuaian habitat dan peta potensi distribusi untuk burung pitta peri. Dalam hal ini, peta kesesuaian habitat dibuat dengan menggunakan fungsi mean() untuk menghitung rata-rata setiap lokasi piksel di semua gambar, dan peta potensi distribusi dibuat dengan menggunakan fungsi mode() untuk menentukan nilai yang paling sering muncul di setiap lokasi piksel di semua gambar.
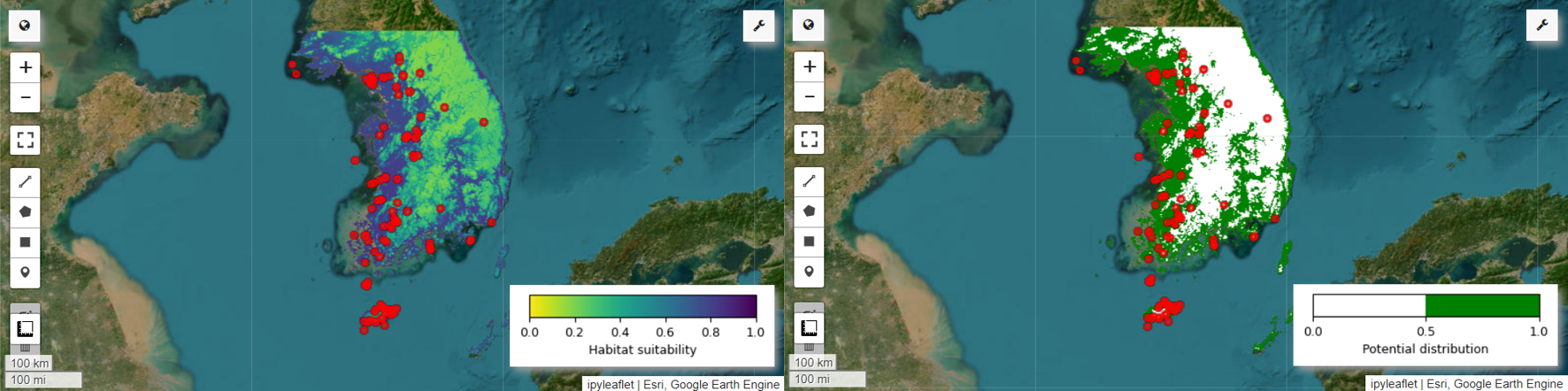
# Habitat suitability map
images = ee.List.sequence(
0, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x))
model_average = ee.ImageCollection.fromImages(images).mean()
Map = geemap.Map(layout={'height':'400px', 'width':'800px'}, basemap='Esri.WorldImagery')
vis_params = {
'min': 0,
'max': 1,
'palette': cm.palettes.viridis_r}
Map.addLayer(model_average, vis_params, 'Habitat suitability')
Map.add_colorbar(vis_params, label="Habitat suitability",
orientation="horizontal",
layer_name="Habitat suitability")
Map.addLayer(data, {'color':'red'}, 'Presence')
Map.centerObject(aoi, 6)
Map
# Potential distribution map
images2 = ee.List.sequence(1, ee.Number(numiter).multiply(4).subtract(1), 4).map(
lambda x: results.get(x)
)
distribution_map = ee.ImageCollection.fromImages(images2).mode()
Map = geemap.Map(
layout={"height": "400px", "width": "800px"}, basemap="Esri.WorldImagery"
)
vis_params = {"min": 0, "max": 1, "palette": ["white", "green"]}
Map.addLayer(distribution_map, vis_params, "Potential distribution")
Map.addLayer(data, {"color": "red"}, "Presence")
Map.add_colorbar(
vis_params,
label="Potential distribution",
discrete=True,
orientation="horizontal",
layer_name="Potential distribution",
)
Map.centerObject(data.geometry(), 6)
Map
Penilaian akurasi dan kepentingan variabel
Random Forest (ee.Classifier.smileRandomForest) adalah salah satu metode pembelajaran ansambel, yang beroperasi dengan membuat beberapa pohon keputusan untuk membuat prediksi. Setiap pohon keputusan belajar secara independen dari berbagai subkumpulan data, dan hasilnya digabungkan untuk memungkinkan prediksi yang lebih akurat dan stabil.
Kepentingan variabel adalah ukuran yang mengevaluasi dampak setiap variabel pada prediksi dalam model Hutan Acak. Kita akan menggunakan importances_list yang ditentukan sebelumnya untuk menghitung dan mencetak rata-rata kepentingan variabel.
def plot_variable_importance(importances_list):
# Extract each variable importance value into a list
variables = [item[0] for item in importances_list]
importances = [item[1] for item in importances_list]
# Calculate the average importance for each variable
average_importances = {}
for variable in set(variables):
indices = [i for i, var in enumerate(variables) if var == variable]
average_importance = np.mean([importances[i] for i in indices])
average_importances[variable] = average_importance
# Sort the importances in descending order of importance
sorted_importances = sorted(average_importances.items(),
key=lambda x: x[1], reverse=False)
variables = [item[0] for item in sorted_importances]
avg_importances = [item[1] for item in sorted_importances]
# Adjust the graph size
plt.figure(figsize=(8, 4))
# Plot the average importance as a horizontal bar chart
plt.barh(variables, avg_importances)
plt.xlabel('Importance')
plt.ylabel('Variables')
plt.title('Average Variable Importance')
# Display values above the bars
for i, v in enumerate(avg_importances):
plt.text(v + 0.02, i, f"{v:.2f}", va='center')
# Adjust the x-axis range
plt.xlim(0, max(avg_importances) + 5) # Adjust to the desired range
plt.tight_layout()
plt.savefig('variable_importance.png')
plt.show()
plot_variable_importance(importances_list)
Dengan menggunakan Set Data Pengujian, kami menghitung AUC-ROC dan AUC-PR untuk setiap proses. Kemudian, kita menghitung AUC-ROC dan AUC-PR rata-rata selama n iterasi.
AUC-ROC merepresentasikan area di bawah kurva grafik 'Sensitivitas (Perolehan) vs. 1-Spesifisitas', yang menggambarkan hubungan antara sensitivitas dan spesifisitas saat nilai minimum berubah. Kekhususan didasarkan pada semua non-kemunculan yang diamati. Oleh karena itu, AUC-ROC mencakup semua kuadran matriks kebingungan.
AUC-PR menunjukkan area di bawah kurva grafik 'Presisi vs. Perolehan (Sensitivitas)', yang menunjukkan hubungan antara presisi dan perolehan saat nilai minimum bervariasi. Presisi didasarkan pada semua kemunculan yang diprediksi. Oleh karena itu, AUC-PR tidak menyertakan negatif sebenarnya (TN).
def print_pres_abs_sizes(TestingDatasets, numiter):
# Check and print the sizes of presence and pseudo-absence coordinates
def get_pres_abs_size(x):
fc = ee.FeatureCollection(TestingDatasets.get(x))
presence_size = fc.filter(ee.Filter.eq("PresAbs", 1)).size()
pseudo_absence_size = fc.filter(ee.Filter.eq("PresAbs", 0)).size()
return ee.List([presence_size, pseudo_absence_size])
sizes_info = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(get_pres_abs_size)
.getInfo()
)
for i, sizes in enumerate(sizes_info):
presence_size = sizes[0]
pseudo_absence_size = sizes[1]
print(
f"Iteration {i + 1}: Presence Size = {presence_size}, Pseudo-absence Size = {pseudo_absence_size}"
)
# Extracting the Testing Datasets
testing_datasets = ee.List.sequence(
3, ee.Number(numiter).multiply(4).subtract(1), 4
).map(lambda x: results.get(x))
print_pres_abs_sizes(testing_datasets, numiter)
def get_acc(hsm, t_data, grain_size):
pr_prob_vals = hsm.sampleRegions(
collection=t_data, properties=["PresAbs"], scale=grain_size
)
seq = ee.List.sequence(start=0, end=1, count=25) # Divide 0 to 1 into 25 intervals
def calculate_metrics(cutoff):
# Each element of the seq list is passed as cutoff(threshold value)
# Observed present = TP + FN
pres = pr_prob_vals.filterMetadata("PresAbs", "equals", 1)
# TP (True Positive)
tp = ee.Number(
pres.filterMetadata("classification", "greater_than", cutoff).size()
)
# TPR (True Positive Rate) = Recall = Sensitivity = TP / (TP + FN) = TP / Observed present
tpr = tp.divide(pres.size())
# Observed absent = FP + TN
abs = pr_prob_vals.filterMetadata("PresAbs", "equals", 0)
# FN (False Negative)
fn = ee.Number(
pres.filterMetadata("classification", "less_than", cutoff).size()
)
# TNR (True Negative Rate) = Specificity = TN / (FP + TN) = TN / Observed absent
tn = ee.Number(abs.filterMetadata("classification", "less_than", cutoff).size())
tnr = tn.divide(abs.size())
# FP (False Positive)
fp = ee.Number(
abs.filterMetadata("classification", "greater_than", cutoff).size()
)
# FPR (False Positive Rate) = FP / (FP + TN) = FP / Observed absent
fpr = fp.divide(abs.size())
# Precision = TP / (TP + FP) = TP / Predicted present
precision = tp.divide(tp.add(fp))
# SUMSS = SUM of Sensitivity and Specificity
sumss = tpr.add(tnr)
return ee.Feature(
None,
{
"cutoff": cutoff,
"TP": tp,
"TN": tn,
"FP": fp,
"FN": fn,
"TPR": tpr,
"TNR": tnr,
"FPR": fpr,
"Precision": precision,
"SUMSS": sumss,
},
)
return ee.FeatureCollection(seq.map(calculate_metrics))
def calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter):
# Calculate AUC-ROC and AUC-PR
def calculate_auc_metrics(x):
hsm = ee.Image(images.get(x))
t_data = ee.FeatureCollection(testing_datasets.get(x))
acc = get_acc(hsm, t_data, grain_size)
# Calculate AUC-ROC
x = ee.Array(acc.aggregate_array("FPR"))
y = ee.Array(acc.aggregate_array("TPR"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_roc = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
# Calculate AUC-PR
x = ee.Array(acc.aggregate_array("TPR"))
y = ee.Array(acc.aggregate_array("Precision"))
x1 = x.slice(0, 1).subtract(x.slice(0, 0, -1))
y1 = y.slice(0, 1).add(y.slice(0, 0, -1))
auc_pr = x1.multiply(y1).multiply(0.5).reduce("sum", [0]).abs().toList().get(0)
return (auc_roc, auc_pr)
auc_metrics = (
ee.List.sequence(0, ee.Number(numiter).subtract(1), 1)
.map(calculate_auc_metrics)
.getInfo()
)
# Print AUC-ROC and AUC-PR for each iteration
df = pd.DataFrame(auc_metrics, columns=["AUC-ROC", "AUC-PR"])
df.index = [f"Iteration {i + 1}" for i in range(len(df))]
df.to_csv("auc_metrics.csv", index_label="Iteration")
print(df)
# Calculate mean and standard deviation of AUC-ROC and AUC-PR
mean_auc_roc, std_auc_roc = df["AUC-ROC"].mean(), df["AUC-ROC"].std()
mean_auc_pr, std_auc_pr = df["AUC-PR"].mean(), df["AUC-PR"].std()
print(f"Mean AUC-ROC = {mean_auc_roc:.4f} ± {std_auc_roc:.4f}")
print(f"Mean AUC-PR = {mean_auc_pr:.4f} ± {std_auc_pr:.4f}")
%%time
# Calculate AUC-ROC and AUC-PR
calculate_and_print_auc_metrics(images, testing_datasets, grain_size, numiter)
Tutorial ini telah memberikan contoh praktis penggunaan Google Earth Engine (GEE) untuk Pemodelan Distribusi Spesies (SDM). Kesimpulan pentingnya adalah fleksibilitas dan kemampuan beradaptasi GEE di bidang SDM. Dengan memanfaatkan kemampuan pemrosesan data geospasial Earth Engine yang canggih, para peneliti dan ahli konservasi dapat memahami dan melestarikan keanekaragaman hayati di planet kita. Dengan menerapkan pengetahuan dan keterampilan yang diperoleh dari tutorial ini, individu dapat menjelajahi dan berkontribusi pada bidang penelitian ekologi yang menarik ini.
 Jalankan di Google Colab
Jalankan di Google Colab
 Lihat sumber di GitHub
Lihat sumber di GitHub